次世代シーケンス(NGS)は、ゲノム解析、トランスクリプトーム解析、メタゲノム解析など、さまざまなアプリケーションで活用されていますが、NGSで得られる膨大なデータの取り扱いは一筋縄ではいきません。
そこで今回は、Python™を使って、比較的簡単にNGSデータの可視化を行う方法についてご紹介いたします。お手持ちのIon Torrent™発現解析データを使って、ぜひ試してみてください。
本ブログでは、準備編に引き続き、Pythonに読み込んだデータの可視化の手順をご紹介します。
※Windows™ PCの利用を想定しています。
▼こんな方におすすめです!
・次世代シーケンシング解析を始めたばかりの方
・コマンドラインのプログラムに興味があるけれど未経験の方
・NGSデータの可視化に興味のある方
データの可視化
準備編でJupyter™ Notebook を使用してIon Torrent™発現解析データの読み込みまでを実施しました。引き続いて、データの中身をさらに見てみましょう。まずは平均値、中央値などの統計量を一通り表示させるdescribeというコマンドを使用してみます。
| df.describe() |

各サンプルについて、上から順に以下の情報が表示されます。
| count | 要素の個数 |
| mean | 平均値 |
| std | 標準偏差 |
| min | 最小値 |
| 25% | 1/4分位数 |
| 50% | 中央値(median) |
| 75% | 3/4分位数 |
| max | 最大値 |
データの分布を箱ひげ図で表示してみます。snsのコマンドでseabornというライブラリを呼び出しています。boxplotで箱ひげ図を指定し、plt.showというコマンドで図を出力します。
| sns.boxplot(data=df) plt.show() |

バイオリンプロットという表示方法もあります。
| sns.violinplot(data=df) plt.show() |

pairplotでは、ペアワイズの散布図がマトリクス上に配置されます。対角線上は同じサンプル同士の組み合わせになり、ヒストグラムが表示されます。
| sns.pairplot(data=df, height=2) plt.show() |

Heatmapで、各サンプルの相関関係を可視化します。数値は各ペアの相関係数を示しています。
| sns.heatmap(df.corr(), square=True, cmap="Blues", annot=True) plt.show() |
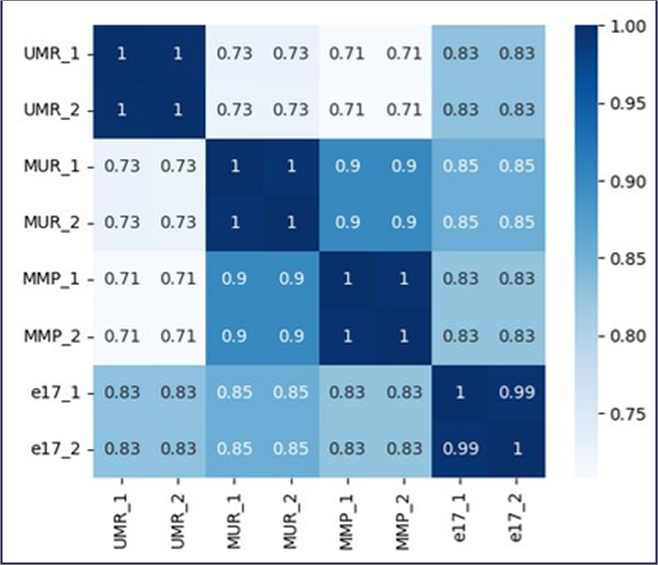
4つのコントロールRNAサンプルをレプリケートで解析していますが、レプリケート同士の相関が非常に高いことがわかります。
最後にscikit-learnというライブラリを使って、主成分分析(principal component analysis)を実施し、プロットを描画してみます。まずデータを標準化し、続いて主成分分析を実施します。ここでは、累積寄与率が90%になるまでの主成分を算出し、printというコマンドで寄与率とともに出力します。最後に、主成分のデータを用いてPCAプロットを作成します。ヒートマップでみたように、各サンプルのレプリケート同士はほとんど重なって分布し、特徴が似ていることがわかります。
| # データの整形 X=df.T sc=preprocessing.StandardScaler() sc.fit(X) X=sc.transform(X) # 主成分分析を実施 pca = sklearn.decomposition.PCA(n_components=0.9) df_pca = pd.DataFrame(pca.fit_transform(X)) df_pca.index = df.columns print(df_pca) print(pd.DataFrame(pca.explained_variance_ratio_)) df_pca['Group'] = ["UMR_1", "UMR_2", "MUR_1", "MUR_2", "MMP_1", "MMP_2", "e17_1", "e17_2"] fig = plt.figure() |

まとめ
- コマンドラインを使った解析で比較的簡単にNGSデータの可視化を行うことができます。
- データをグラフで表示することで、各サンプルのデータの概要をより直感的に理解することができます。
- このほかにもPythonにはさまざまなNGSデータ解析ツールが備わっており、活用することでNGSデータを理解し、その生物学的意味の考察に役立てることができます。
Ion Torrent™次世代シーケンスのデータを可視化して理解に役立てたい、論文等でわかりやすい図を作成したい、Pythonなどのツールを活用した解析をやってみたいが具体的にどうしたらよいかわからない、といった場合には、ぜひ弊社テクニカルサポートのデータ解析コンサルティングサービスをご検討ください。解析手法のレクチャーやお手持ちのデータを使った受託解析まで、幅広いご相談に対応いたします。
次世代シーケンサ(NGS)入門
次世代シーケンスの原理や何ができるかがよくわからない、または自分の研究領域にどのように活用できるかわからないという方向けに、次世代シーケンスの基本や各研究領域に特化したアプリケーションをまとめました。リンク先から、それぞれの領域に応じたページをご覧いただけます。
Python is a trademark of Python Software Foundation.
Windows is a trademark of Microsoft Corporation.
Jupyter is a trademark of NumFOCUS, Inc.
研究用にのみ使用できます。診断用には使用いただけません。