次世代シーケンサでの発現解析に取り組む際、RNAサンプルが「少なすぎる」「分解している」という状況は珍しくありません。特にFFPE(パラフィン包埋切片)由来サンプルや、貴重な臨床検体では、収量が限られている、あるいは品質が非常に低下しているケースも多々あります。
こうした「難易度の高いサンプル」でも信頼できるトランスクリプトームデータを得たい、これは研究者にとって切実な課題ではないでしょうか。
そこで今回は、RNA-SeqとIon AmpliSeq™ Transcriptomeという2つの代表的な手法を用いて、微量・分解RNAでどこまで解析可能なのかを実際に試してみました。
合わせて読みたい:低発現遺伝子にフォーカスしたIon AmpliSeq RNAテクノロジーによるNGS発現解析
▼こんな方におすすめです!
- 次世代シーケンサを用いた発現解析に興味がある
- 量が少ないサンプルでの発現解析を検討している
- 長期保存などにより、分解の進んでしまったサンプルの発現解析を検討している
材料と方法
材料:
- HeLa細胞由来RNA(※花王株式会社さまより提供)
RNAサンプルは、95 ℃に設定したApplied Biosystems™ ProFlex™ PCR Systemにセットし、1時間または2時間の過熱処理を行うことで、段階的に分解を進行させたRNAを調整した。RNAの分解度評価には、Agilent™社製BioAnalyzer™システムを用いた。
Ion AmpliSeq Transcriptome:
- Invitrogen™ SuperScript™ VILO™ cDNA Synthesis Kit(製品番号:11754050)
- Ion AmpliSeq™ Transcriptome Human Gene Expression Panel, Chef-Ready Kit(製品番号:A31446)
RNA-Seq:
- Invitrogen™ Low Input RiboMinus™ Eukaryote System v2(製品番号:A15027)
- Ion Torrent™ Ion Total RNA-Seq Kit v2(製品番号:4475936)
- Ion Xpress™ RNA-Seq Barcode 1-16 Kit(製品番号:4475485)
(必要試薬の詳細は各ユーザーガイドをご参照ください)
使用機器・ソフトウエア:
- Ion GeneStudio™ S5 Prime System(製品番号:A38196)
- Ion Chef™ システム(製品番号:4484177)
- Torrent Suite™ Software Version 5.18.1
方法:
各ユーザーガイドに準拠してライブラリー調製・シーケンスを実施
推奨RNAインプット量:
- RNA-Seq:100 ng
- Ion AmpliSeq Transcriptome:10 ng
解析ツール:
- RNA-Seq:RNASeqAnalysisプラグイン(Torrent Suite Software)
- Ion AmpliSeq Transcriptome:Ion AmpliSeq™ RNAプラグイン(Torrent Suite Software)
- R version 4.2.3
結果と考察
■微量RNAにおけるライブラリー調製
ライブラリー調製結果を表1に示します。RNA-Seqでは、RNA投入量が100 ngの場合、分解が進んだRNAであってもライブラリー調製が可能でした。しかし、RNA投入量を100 pg(約10細胞相当)まで減らした場合には、RNAの品質に問題がなくてもライブラリーを得ることはできませんでした。一方、Ion AmpliSeq Transcriptomeでは、いずれのRNA投入量においても、次世代シーケンサ解析に十分な濃度のライブラリーを得ることができました。
表1. ライブラリー作製条件とその成否
| 手法 | RNA インプット量 | 分解度 | ライブラリー 作製成否 |
|---|---|---|---|
| Ion AmpliSeq Transcriptome | 100 pg | コントロール | 〇 |
| 10 ng | コントロール | 〇 | |
| 10 ng | DV200 = 50-60% | 〇 | |
| 10 ng | DV200 = 30-40% | 〇 | |
| RNA-Seq | 100 pg | コントロール | ✕ |
| 100 pg | DV200 = 50-60% | ✕ | |
| 100 pg | DV200 = 30-40% | ✕ | |
| 100 ng | コントロール | 〇 | |
| 100 ng | DV200 = 50-60% | 〇 | |
| 100 ng | DV200 = 30-40% | 〇 |
DV200:200 nt 以上のRNA断片の比率
■分解RNAにおけるライブラリー調製
RNA-Seq、Ion AmpliSeq Transcriptomeのいずれにおいても、今回準備した分解度のRNAでは全てライブラリー調製が可能でした。
■ライブラリー調製の成功率の差を生む要因
RNA-SeqはビーズによるrRNA除去、RNA断片化、cDNA合成、アダプター付加など多段階の工程を経て最終的にPCRでライブラリーを増幅します。そのため、インプット量が少ない場合には酵素反応の効率低下や各工程でのサンプルロスが起こりやすく、ライブラリー調製が困難になると考えられます。
一方でIon AmpliSeq TranscriptomeはcDNA合成直後にマルチプレックスPCRでターゲット遺伝子を増幅します。このため、以降の工程で十分なDNA量を確保しながら反応を進めることができ、微量・分解の進んだRNAからでも安定してライブラリー調製が可能です。さらにrRNA除去の前処理が不要であることも、サンプルロスを防ぎ、高い実験成功率につながる要因と考えられます。
■微量RNAでの遺伝子検出率
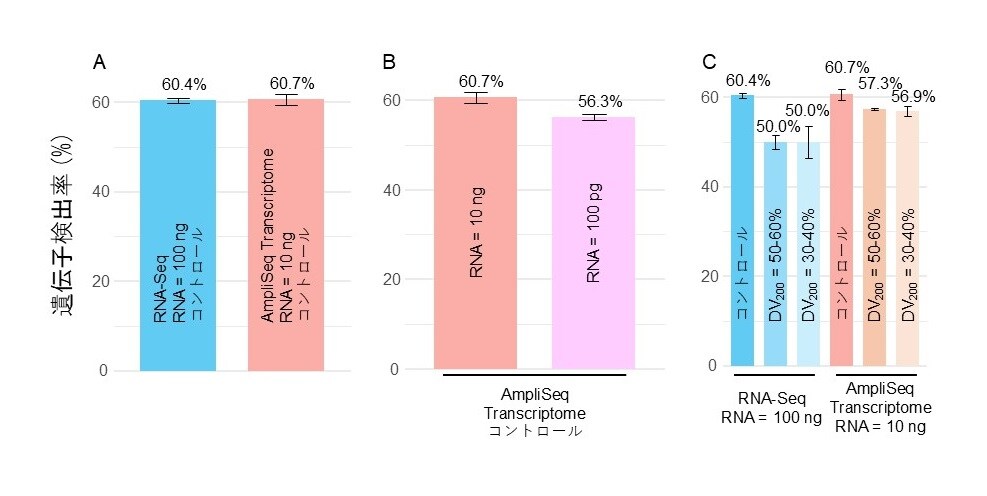
次に各手法で微量RNAを用いた際にどの程度の遺伝子検出率を維持できるか検証しました。図1. A~Cでは、Ion AmpliSeq Transcriptomeで解析対象としている約2万遺伝子のうち、10リード以上が得られた遺伝子の割合を実験条件ごとに示しています。
まず、推奨インプット量(RNA-Seq = 100 ng、Ion AmpliSeq Transcriptome = 10 ng)で比較(図1. A)すると、検出率はそれぞれ60.7%と60.4%であり、検出できた遺伝子数に大きな差は認められませんでした。
さらに、100 pg(約10細胞相当)のRNA量でのIon AmpliSeq Transcriptomeの結果(図1. B)は、同10 ng条件には劣るものの50%以上(約1万種類)の遺伝子を検出できました。
RNA-Seqでは100 pgのRNA量においてそもそもライブラリーを調製することも困難でしたが、Ion AmpliSeq Transcriptomeであれば十分な解析結果が期待できることが示されました。
■分解RNAでの遺伝子検出率
RNAの分解度が遺伝子検出率に与える影響についても比較解析しました(図1. C)。
RNA-SeqではDV200 = 50~60%および30~40%のいずれの条件でも検出率は約50%にとどまりました。一方、Ion AmpliSeq TranscriptomeではDV200 = 50~60%では57.3%、30~40%では56.9%と、RNA-Seqを上回る遺伝子検出率を維持できました。
Ion AmpliSeq TranscriptomeのユーザーガイドではDV200 > 30%が推奨されており、分解が進んだサンプルでも約150 bpのターゲット領域を増幅できる限り良好な結果が得られることが裏付けられています。
■遺伝子検出率の差を生む要因
これらの結果はライブラリー調製原理の違いに起因すると考えられます。
Ion AmpliSeq TranscriptomeはcDNA合成後に約150 bpのターゲット領域をマルチプレックスPCRで直接増幅し、下流工程に必要なDNA量を確保することで、微量RNAでも安定したライブラリー調製が可能です。これにより、100 pgといった極めて少量のRNAからでも約1万種類の遺伝子を検出できたと考えられます。
同じ原理により、分解RNAの場合も解析に必要な断片が残っていれば高感度に検出でき、DV200が30%程度でも比較的高い検出率を維持できます。
しかしRNA-Seqの場合、先述の原因により微量RNAはライブラリーが調整できませんでした。またRNAが分解している際はライブラリー調製自体を行うことはできるものの、
- rRNA除去時にmRNAを効率的に精製できない
- ライブラリー調製時の断片化工程で過剰に分断される
などにより解析対象となる遺伝子数が低下してしまいます。
■Ion AmpliSeq Transcriptomeは分解RNAでも再現性のある実験が可能
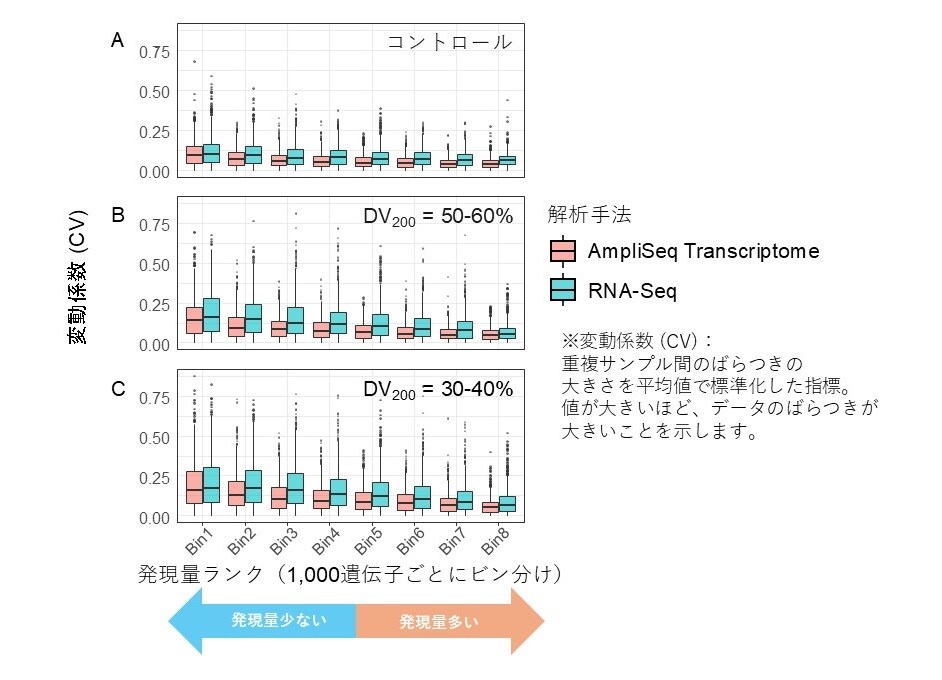
最後に各手法における重複サンプル間の再現性を比較しました(図2. A~C)。図2.では遺伝子を発現量ごとに分類し、各群におけるサンプル間のばらつきを評価しています。
コントロールサンプル(図2. A)では、いずれの手法も発現量が低い遺伝子ほどばらつきが大きい傾向が確認されました。しかし、同一の発現量ランクで比較すると、Ion AmpliSeq TranscriptomeはRNA-Seqよりもばらつきが小さい結果となりました。
また分解RNAサンプル(図2. B、C)では、どの手法でも分解が進むにつれてばらつきが増加する傾向が見られましたが、Ion AmpliSeq TranscriptomeはRNA-Seqに比べてばらつきが小さく、より安定したデータを提供できることが示されました。
また分解RNAサンプル(図2. B、C)では、どの手法でも分解が進むにつれてばらつきが増加する傾向が見られましたが、Ion AmpliSeq TranscriptomeはRNA-Seqに比べてばらつきが小さく、より安定したデータを提供できることが示されました。
まとめ
今回の実験では微量あるいは分解の進んだRNAサンプルを用いて次世代シーケンサでの遺伝子発現解析を行い、その結果を比較検討しました。次世代シーケンサでの解析はサンプル当たりのコストも決して安価ではないため、状態のよくないサンプルで解析を進めるのには経済的にもリスクが伴います。そのため今回の検証結果が普段の実験手法の選択のご判断に少しでもお役に立てば幸いです。ただし、実際のサンプルでは異なる結果が出る可能性もありますので、その点も考慮しながら参考にしていただければと思います。
テクニカルサポートお問い合わせ先
メール:jptech@thermofisher.com
電話:03-4520-5288(ダイレクト番号…NGS実験系3番)
皮脂RNAモニタリング®技術についての紹介動画
花王株式会社では、あぶらとりフィルム一枚で採取した皮脂中にヒトに由来するmRNAが存在することを発見しました。皮脂中のヒトmRNA発現量の網羅解析にはIon AmpliSeq Transcriptome技術が応用されています。井上高良氏(花王株式会社)の皮脂RNAモニタリング技術紹介動画を公開中です。技術詳細と研究事例をわかりやすく解説している動画をご覧ください。
研究用にのみ使用できます。診断用には使用いただけません。