世界各地で新型コロナウイルス(SARS-Cov-2)のゲノム配列が解読され、公共データベースに登録されています。公開された配列データを利用するだけでなく、自施設で解読したウイルスゲノム配列と合わせて系統解析を行うことで、ウイルスの感染伝播の追跡や、サンプルの系統情報の把握に役立つ情報が得られます。以前、弊社のブログ“ウイルス配列の入手とアライメント”にて、公開されている配列の取得とアライメントの方法についてご案内しました。
今回は、アライメントしたファイルを用いたウイルス配列の系統解析についてご紹介いたします。
▼こんな方におすすめです!
・自分の施設で解読したウイルスの配列と既報の配列を比較したい
・ウイルス配列の系統関係を調べたい
なお、IonTorrentシーケンサで新型コロナウイルスゲノムを解析できる、Ion Torrent™ Ion AmpliSeq™ SARS-CoV-2 Research Panelの内容をこちらのサイトで確認できます。
データの準備とマルチプルアライメント
2020年10月時点で、新型コロナウイルスのゲノム配列情報を入手できるデータソースGISAIDには117,693配列が、NCBI virusには25,399配列が登録されています。日本でも新型コロナウイルス感染症に関する研究データのポータルサイト「COVID-19データポータルJAPAN」が公開され、このサイトからCOVID-19に関連する配列情報検索サービスDDBJ ARSAを経由して配列の取得が可能です。
DDBJ ARSAでは“Search Condition”メニューからさまざまな条件で配列の絞り込みが可能です。たとえば、“Definition”の項目で”complete genome”を選択して全長配列データを絞り込んだり、“Feature” の項目で“country=Japan” を指定して日本で採取された検体由来の配列だけをダウンロードすることもできます。
マルチプルアラインメントには以前のブログでご紹介したUnipro UGENEというオープンソースのソフトウェアのほかに、MAFFTというアライメントツールのオンラインバージョンも使用できます1。データベースからダウンロードしたfasta形式のファイルをアップロードし、submitボタンをクリックすると解析が始まります。メールアドレスを登録しておくと解析の開始と完了時にメールが届く仕組みです。
系統解析の実施
マルチプルアラインメントが完了したら、系統解析を実行します。最尤法を用いて系統樹探索を行うRAxMLというプログラムのオンラインバージョンを利用できます2。アライメント済みのfastaファイルをアップロードし、submitをクリックすると解析が始まります。ブートストラップ値(作成した系統樹の信頼性を評価する値)を得たい場合はbootstrappingにチェックを入れてから開始します。こちらもメールアドレスを登録しておくと解析完了時にメールが届きます。約3万塩基対の長さのある新型コロナウイルスゲノム配列を800検体分ほどアライメントしたファイルをsubmitした場合、完了まで20時間ほどかかります。また、データが大きすぎる場合は解析がタイムアウトすることもありますのでご注意ください。
系統樹の確認と編集
系統樹が得られたら樹形を確認します。系統解析の結果はnwkというファイル形式で出力されることが多いです。このファイルをFigTreeというオープンソースのソフトウェアで開くと、系統樹の閲覧だけでなく、有根・無根系統樹の選択、注目する配列やクラスターの色分けなどの編集が行えます。
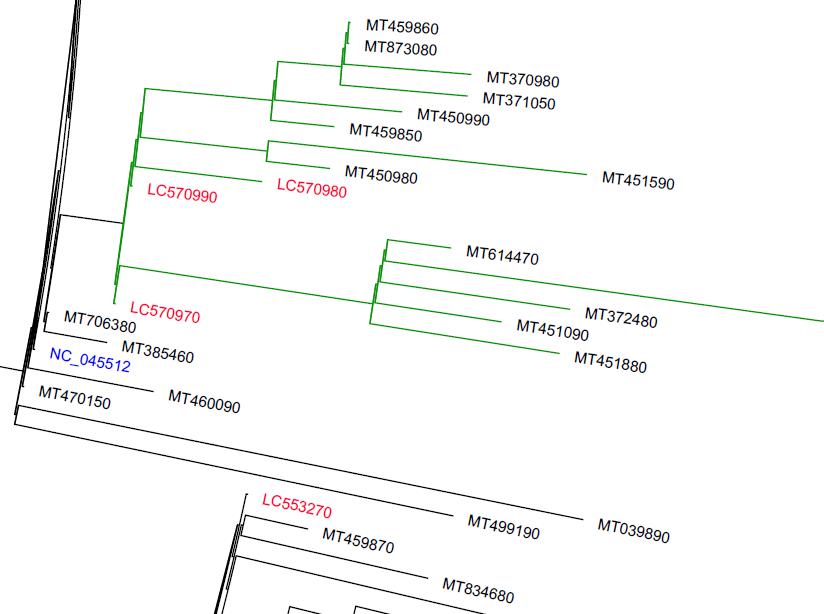
Figtreeを用いて編集した系統樹の一部を図に示します。各ウイルスゲノム配列は登録アクセッション番号で示されています。日本の施設から登録された配列を赤字で、それらの配列を含む群の枝を緑で、また参照配列として使用した配列(NC_045512)を青字で表示するなど、注目したい配列を色分けして結果をわかりやすく示すことができます。
まとめ
・新型コロナウイルスの登録配列数は日々増加し続けています。
・公共データベースから得られる配列を使って、オープンソースのソフトウェアやオンラインのプログラムを利用し、どなたでも簡単に系統解析を行い、結果を確認できます。
・弊社Ion Torrent™ Ion AmpliSeq™SARS-Cov2-panel等を使用して決定した新型コロナウイルスのゲノム配列を加えて、既存の配列との違いを確認し、その関係性を調べることが可能です。
・新型コロナウイルスに関連するソリューションはこちらのページをご覧ください。
・NGS等のデータ解析でお困りの方は、データ解析コンサルティングサービスをご検討ください。
次世代シーケンス データ解析オンライン個別相談
IonTorrentユーザでデータ解析に関する具体的な課題をお持ちの方には、無料の個別相談の時間を開設しています。
References
1. Katoh K, Rozewicki J, Yamada KD. MAFFT online service: multiple sequence alignment, interactive sequence choice and visualization. Brief Bioinform. 2019 Jul 19;20(4):1160-1166. doi: 10.1093/bib/bbx108.
2. Kozlov AM, Darriba D, Flouri T, Morel B, Stamatakis A. RAxML-NG: a fast, scalable and user-friendly tool for maximum likelihood phylogenetic inference. Bioinformatics. 2019 Nov 1;35(21):4453-4455. doi: 10.1093/bioinformatics/btz305.
*ここに記載したソフトウェアやデータベースの一部は、弊社の製品サポートの対象ではありません。それぞれのソフトウェアの詳細機能は、開発元やマニュアルにてご確認ください。
研究用にのみ使用できます。診断用には使用いただけません。



