DNAマイクロアレイは登場してからすでに20年がたち、熟成した技術といえます。他方、近年、次世代シーケンサ(NGS)の性能が向上するにつれて遺伝子発現解析をNGSで行うRNA-Seqも広く用いられるようになってきました。しかしマイクロアレイは次世代シーケンサに比べて解析の容易さ、コスト的なメリットなどから、まだまだ強力な網羅的な遺伝子発現解析ができるとして、近年見直されてきている技術でもあります。弊社でも、マイクロアレイと次世代シーケンサ、両方の特長を生かして相乗的に利用していくことをお勧めしています。
そこでこのブログでは、次世代シーケンサに比べて、マイクロアレイの良さはどこにあるのかをご紹介します。
▼もくじ
マイクロアレイは解析が簡単!~バイオインフォマティクスに詳しくなくてもTACで簡単解析
マイクロアレイの解析は、次世代シーケンサと比べて圧倒的に簡単なことが、大きな特長です。ここでは、そのユーザーフレンドリーなマイクロアレイ解析について四つの大きなポイントをご紹介します。
1. マイクロアレイはデータサイズが小さく、扱いやすい
2. ソフトウエアに任せれば、解析結果が出るまで数秒~数分
3. バイオインフォマティクスに詳しくなくても解析ができる
4. 選択的スプライシングの解析が簡単に行いやすい
1. マイクロアレイはデータサイズが小さく、扱いやすい
次世代シーケンサのデータは圧縮状態でもGB(ギガバイト)レベルのビッグデータで、多くのケースで別途サーバーが必要となります。しかしマイクロアレイのデータはMB(メガバイト)レベルです。生データ(解析に使うCELファイル)をご使用のパソコンに入れることができますので、サーバーを用意する必要はありません。サーバーを用意するだけでコストがかかりますし、パソコンで手軽に解析ができるというのは大きなメリットです。
2. ソフトウエアに任せれば、解析結果が出るまで数秒~数分
弊社のマイクロアレイ専用の無償ソフトウエアTranscriptome Analysis Console(TAC)を用いて、発現解析の代表的なアレイGeneChipTM Clariom Sを解析する場合、数秒から数分というわずかな時間で解析結果を入手できます※。一方、次世代シーケンサの解析には一般的に数日から数週間が必要と言われています。
※推奨以上のパソコンスペックであれば、Clariom S でN=2、2群の解析であれば30秒から1分程度で解析結果を出すことができます。
3. バイオインフォマティクスに詳しくなくても解析ができる
TACは生物学研究者向けに開発されたソフトウエアで、バイオインフォマティクスの詳しい知識がなくても解析ができるようにデザインされています。使い方は直感的でわかりやすく、数値化された生データ(CEL)ファイルを読み込ませ、解析ボタンを押すだけでデータQCや遺伝子発現解析の結果を確認できます。TACの使い方についてはブログや動画でご紹介していますので、その使いやすさをぜひ実感してみてください。
なお、次世代シーケンサでは複数のソフトウエアからなるパイプラインを利用する必要があり、多くの場合はバイオインフォマティクスの知識を必要とします。
4. 選択的スプライシングの解析が簡単に行いやすい
Clariom Dなどの選択的スプライシングの解析ができるマイクロアレイでは、TACを用いて選択的スプライシングの解析がわかりやすいビジュアルで行えます。
下図は、Clariom DのデータをTACで解析した結果です。LiverとMuscleサンプルの各エクソンの発現量が表示されています。青は発現量が多く、黄色は発現量が小さいことを意味しています。
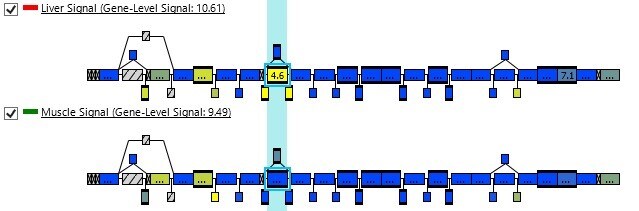
上図では選択されているエクソン領域において、Liverでは黄色(=発現量が小さい)、Muscleでは青色(=発現量が大きい)に表示されています。つまり、Liverにおいて黄色のエクソンがスプライスアウトされている可能性が高いことが推測されます。
マイクロアレイとTACの組み合わせであれば、ビジュアルでもわかりやすく選択的スプライシングの解析ができます。また、解析結果を出すまでのスピードも非常に速く、Clariom DでN=2、2群であれば30分程度※で上図の結果を出すことができます
※パソコンのスペックや環境による。
このように、マイクロアレイでは次世代シーケンサと比べてデータが軽くて扱いやすく、短時間で結果を出せて、バイオインフォマティクスに詳しくなくても解析が可能です。試しにTACを使ってみたい!という方は、弊社のWEBからサンプルデータをダウンロードして解析いただけます。サンプルデータのダウンロード方法はこちらをご覧ください。
マイクロアレイでは、RNA-Seqをしのぐ精度で低発現遺伝子もしっかり解析
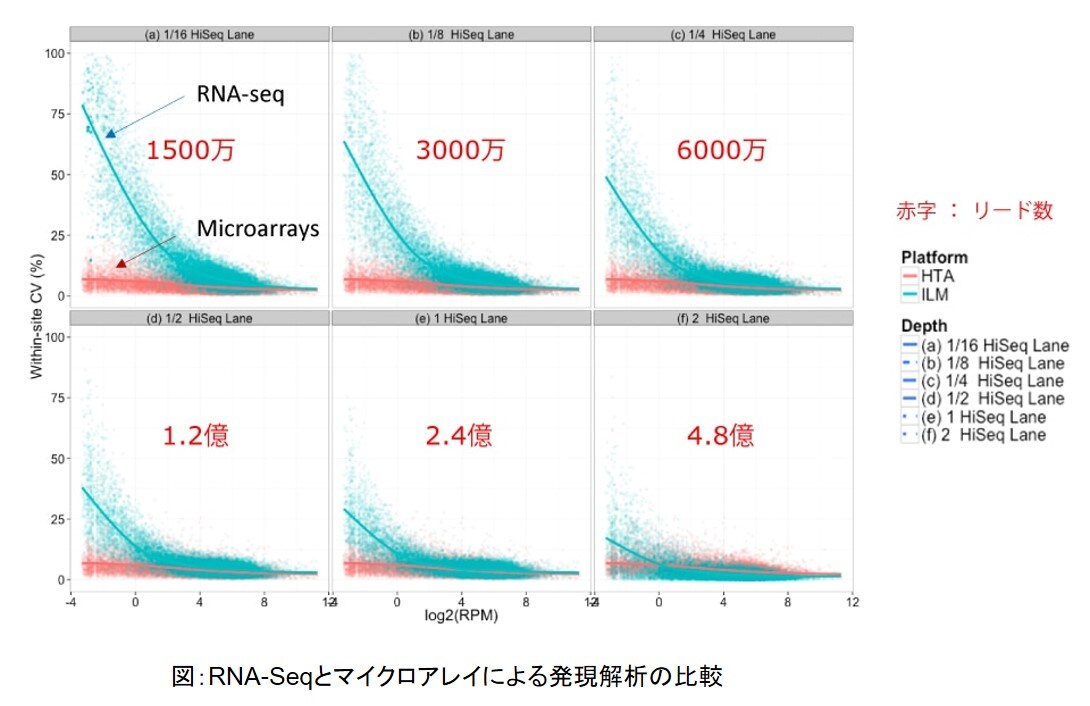
マイクロアレイにはRNA-Seqのようにリード数という概念がありません。しかしRNA-Seqではリード数によってその精度に違いが生じます。RNA-Seqとマイクロアレイの発現精度の違いとして、下図のようなデータがあります。
※Xu, W., et al. Human transcriptome array for high throughput clinical studies. Proc Natl Acad Sci USA 108(9):3707–3712 (2011).
上図の横軸は遺伝子の発現量を、縦軸は測定値のばらつきの大きさを表しています。
図の左上、1500万と赤字で書かれているグラフに注目してください。この赤字はRNA-Seqのリード数を示しています。マイクロアレイは低発現の遺伝子でも測定値のばらつきが小さいのに対し、RNA-Seqでは発現量が低くなるにつれて測定のばらつきがどんどん大きくなっていることがわかります。このように、リード数が少ない条件でのRNA-Seqでは低発現遺伝子の精度がどうしても落ちてしまいます。RNA-Seqで低発現遺伝子を高精度で解析する場合はリード数を増やしていく必要があります。図の右下のように4.8億リードまでリード数を増やすことにより、ようやくマイクロアレイと同程度の解析性能を持つことがわかります。一般的に行われているRNA-Seqではリード数にして4000万程度で解析することが多いようです。上述したとおり、低発現の遺伝子まで精度よく測定するためには少なくともこの10倍以上のリード数が必要で、コストがかかります。
RNA-Seqでしか得られない情報が必要であればこのコストは必要経費として割り切れますが、もしもデータベース上にある既存遺伝子の解析が主な目的であるのならば、Clariom Dなどのマイクロアレイを用いた方がはるかに高精度かつ低コストで解析ができるのです。
マイクロアレイは安価で解析ができるアプリケーション
次世代シーケンサの場合はシステムやリード数によってランニングコストは異なりますが、一般的に次世代シーケンサよりもマイクロアレイの方が安価で解析が可能です。
弊社の代表的なマイクロアレイとして、Applied Biosystems™ Clariom DとApplied Biosystems™ Clariom Sのランニングコストをご紹介します。

※試薬・マイクロアレイ、ハイブリ洗浄染色試薬などすべて含む価格です。価格は2020年10月現在のもので、機器が別途必要です。
Clariom Dは全転写産物が解析できるトランスクリプトームレベルの解析ができるアレイ、Clariom Sは遺伝子発現に特化したアレイです。マイクロアレイ登場当時の価格をご存じの方であれば、驚異的なまでにお安くなりました。
なお、受託解析の場合は会社により価格が異なりますので、各社にお問い合わせください。
まとめ
マイクロアレイは熟練した技術ではありますが、次世代シーケンサと比べて解析の簡単さ、低発現領域解析の精度の高さ、コストの安さなどから、最近また見直されてきている技術です。このブログではマイクロアレイに焦点を当ててご紹介しましたが、もちろん次世代シーケンサでしかできない解析(未知遺伝子の発見やゲノム配列の解読など)や次世代シーケンサを使うメリットもあります。両者の特長に応じて使い分けをしていくことをお勧めします。
マイクロアレイの手引き
マイクロアレイの原理や基礎、ワークフローなどについて学べるページです。
研究用にのみ使用できます。診断用には使用いただけません。



