キャピラリーシーケンサでのランが終わり、いざシーケンスデータ(.ab1)を開いてみると、波形は確認できるものの、ベースコールされた塩基配列が”NNNNN”だったことはありませんか?
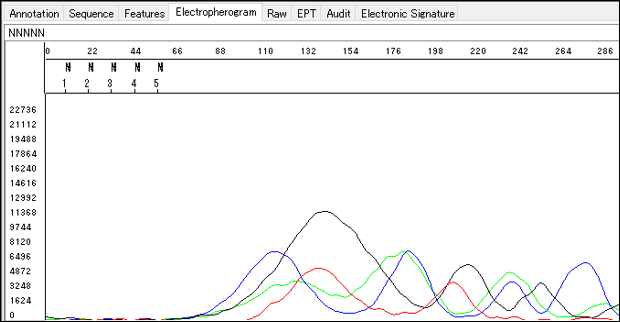
図1: ”NNNNN”
これはソフトウエアを用いて再度解析することによって、ベースコールし直すことができる場合があります。
そこで今回は、”NNNNN”となっているサンプルファイルを再解析する方法について、ご紹介いたします。
▼こんな方におすすめです!
- サンガーシーケンスを実施している方
- サンガーシーケンスデータのトラブルシュートに興味をお持ちの方
”NNNNN”となってしまう原因は?
正しくベースコールできていない場合、生データ「Raw」を見てみましょう。
- Sequencing Analysisソフトウエアの場合、「Raw」タブをクリックします。
- Sequencing Analysisソフトウエアを開く
- 「File」 > 「Add Sample(s)…」をクリック
- 任意の.ab1ファイルを選択し、「Add Selected Samples >>」 > 「OK」をクリック
- 該当のサンプルファイルの「Show」にチェックをつける
- 「Raw」タブをクリック

図2:Sequencing Analysisの場合のRaw
- Thermo Fisher™ Connect™ PlatformのQuality Checkの場合、1つのサンプルファイルをダブルクリックし、「Raw」タブをクリックします。
- Thermo Fisher Connect Platformにサインイン(https://apps.thermofisher.com/)
- 「Quality Check」 Appをクリック
- 「Create New Project」を選択後、「Type a project name」にプロジェクト名を入力し、「OK」をクリック
- 「Import From」 > 「My Computer」をクリック
- 任意の.ab1ファイルを選択し、「Open」をクリック
- 左枠の「QC」をクリック
- 「Import Project Settings」で「No」を選択し、「Continue」をクリック
- 該当の1サンプルをダブルクリック
- 「Raw」タブをクリック

図3:Quality Checkの場合のRaw
ターゲット配列が短いものの、長いターゲット用のランモジュールでランしているために、正しく解析できずに、解析不能”NNNNN”となっている場合が多いです。
この場合、短いターゲット用のランモジュールでランをすれば解消することがあります。
それでも解消しない場合や既に取得したデータファイルについては、以下の手順で再解析を試してみましょう。
Sequencing Analysis ソフトウエアでの再解析
- 手順1: Rawデータにて、最後のピークの位置を確認
- Rawデータの波形の上にカーソルを移動し、ドラッグすると、十字の点線が表示されます。
- ドラッグしたまま、最後のピークに移動し、横軸(データポイント)の数字を確認します。
- 図4では最後のピークのデータポイントは「2548」です。

図4:SeqAでのデータポイントの確認
- 手順2:「Stop」に確認したデータポイントを入力
- 該当サンプルの「Stop」に手順1で確認した最後のピークのデータポイントを入力します。
- 「Stop」はRawデータの解析終了ポイントですので、これで解析終了位置を指定し直すことになります。
- 図4の結果であれば、「Stop」に「2548」を入力します。
- 手順3:再解析の実行
- 該当のサンプルの「BC」にチェックをつけ、緑の三角ボタン(「Start」 > 「Start Analysis」)をクリックし、再解析を実行します。
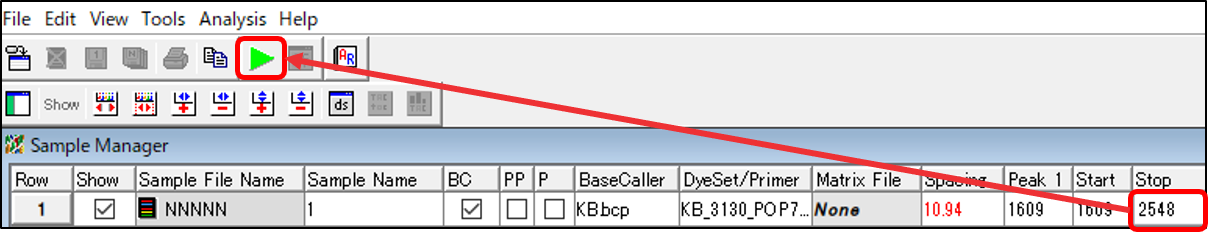
図5:SeqAでの再解析の実行

図6:SeqAでの再解析後画面
再解析後、「Electropherogram」タブをクリックし、波形とベースコールされた塩基を確認します。
再解析したファイルは「File」 > 「Save Sample(s)」で上書き保存することができます。
- 該当のサンプルの「BC」にチェックをつけ、緑の三角ボタン(「Start」 > 「Start Analysis」)をクリックし、再解析を実行します。
Quality Check Appでの再解析
- 手順1:サンプルファイルの選択
- 「Traces」画面にて、再解析したいファイルのチェックボックスにチェックをつけます。
- 手順2:再解析条件の指定
- 「Basecall」をクリックします。
- 「Ending base and Calling Ns」をクリックし、「At PCR stop」のチェックボックスにチェックを付けます。
- 手順3:再解析の実行
- 「Analyze」をクリックし、再解析を実行します。

図7:QCでのサンプル再解析の実行

図8:QCでの再解析後画面
再解析後、サンプルをダブルクリックし、波形とベースコールされた塩基を確認します。
解析したファイルは以下の手順で出力することができます。
- 「Actions」 > 「Export Traces」画面にて、「*.ab1」にチェック > 「OK」をクリック
- 「Analyze」をクリックし、再解析を実行します。
まとめ
- 短いターゲットを長いランモジュールで泳動すると”NNNNN”となることがある
- 生データ(Raw)を確認することで原因を推測できる
- ソフトウエアで再解析することで改善できる場合がある
“NNNNN”となったデータがありましたら、上記の手順をぜひお試しください。
関連記事:サンガーシーケンスの原理
【無料公開中】サンガーシーケンスの基礎
このケミストリーガイドでは、ワークフローからアプリケーションまでサンガーシーケンスの基本原理を学べます。
研究用にのみ使用できます。診断目的およびその手続き上での使用はできません。



