さて、第4回です。第3回に引き続き、実例を使ってベクターマップ解読の流れをご説明します。今回は哺乳類細胞以外の発現系用ベクターの例をご紹介します。
„Åæ„ÅöÊúÄÂàù„Å´„Éô„ÇØ„Çø„ɺ„Éû„ÉÉ„Éó„Å´Âê´„Åæ„Çå„Çã„Ç®„ɨ„É°„É≥„Éà„Å´„ŧ„ÅфŶ„Åß„Åç„Çã„ÅÝ„ÅëÁ∞°Âçò„Å´„Åæ„Å®„ÇÅ„ÅüË≥áÊñô„Çí˺â„Åõ„Åæ„Åô„ÅÆ„ÅßÂÆü‰æã„Å®‰∏ÄÁ∑í„Å´„Åì„Å°„Çâ„Çí˶ã„Ŷ„ÅÑ„Åü„ÅÝ„Åè„Å®„Çà„Çä„Çè„Åã„Çä„ÇÑ„Åô„ÅÑ„Å®ÊÄù„ÅÑ„Åæ„Åô„ÄÇ„ÇÇ„ÅóÊÉÖÂݱ„ÅåË∂≥„Çä„Å™„ÅÑÂÝ¥Âêà„ÅØÊúÄÂæå„Å´ÈÅéÂ骄Éñ„É≠„Ç∞„ÅÆ„É™„É≥„ÇØ„Çí„Åæ„Å®„ÇÅ„Åæ„Åó„Åü„ÅÆ„Åß„ÄÅ„Åì„Å°„Çâ„ÇÇ„ÅîÂèÇÁÖß„Åè„ÅÝ„Åï„ÅÑ„ÄÇ
▼もくじ
- ベクターマップに含まれるエレメントについて
- 例1:一般的な大腸菌発現用ベクター(N末端タグ付き)
- 例2:一般的な大腸菌用発現ベクター(N末端タグなし、C末端タグ付き)
- 例3:昆虫細胞発現で使うバキュロウイルス作成用ベクター(Bac-to-Bac Baculovirus Expression System用)
- 例4:酵母Pichia発現用ベクター
- ÊúÄÂæå„Å´
ベクターマップに含まれるエレメントについて
過去ブログで何度かご説明済みですがより簡単にまとめてみました。
‚ëÝÔΩû‚먄ÅÆË™¨ÊòéÔºö
‚ëݧßËÖ∏ËèåÂÜÖ„ÅßÊ©üËÉΩ„Åô„Çãorigin
②大腸菌内で機能する抗生物質耐性遺伝子
③目的遺伝子発現用プロモーター
④クローニングサイト
⑤融合タグ配列(この例はN末端タグ + プロテアーゼ切断配列)
‚ë•„Éù„É™A‰ªòÂäÝ„Ç∑„Ç∞„Éä„É´
⑦シーケンス用プライマーのプライミングサイト
⑧大腸菌以外の宿主用選択マーカー(主に抗生物質耐性遺伝子)
⑨プロモーター(⑧用)
‚ë©„Éù„É™A‰ªòÂäÝ„Ç∑„Ç∞„Éä„É´Ôºà‚ëßÁî®Ôºâ
⑪大腸菌用プロモーター(⑧用)
⑫機能上不要な配列(f1 ori)
⑬発現調節エレメント
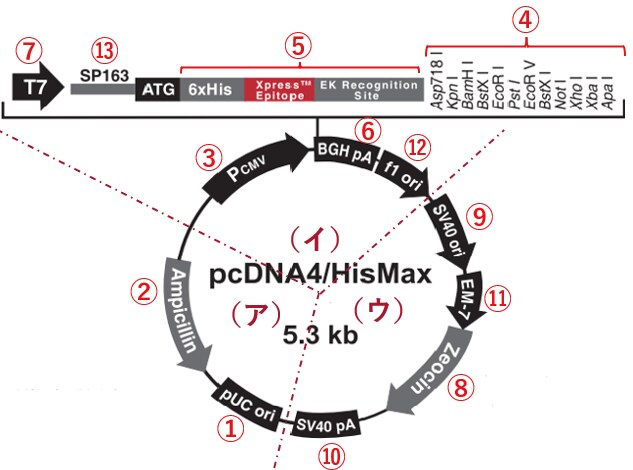
例1:一般的な大腸菌発現用ベクター(N末端タグ付き)
ÁõÆÁöÑÈÅ∫‰ºùÂ≠êÔºàGOIÔºâ„Çí§ßËÖ∏Ëèå„ÅߧßÈáèÁô∫Áèæ„Åï„Åõ„Çã„Åü„ÇÅ„Å´„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÇíÂÖ•Êâã„Åó„ÄÅ„Éû„ÉÉ„Éó„Å®„Éû„Éã„É•„Ç¢„É´„Çí˶ã„Ŷ‰Ωø„ÅÑÊñπ„ÇíËÄÉ„Åà„Ŷ„ÅÑ„Çã„Å®„ÅÑ„ÅÜÁä∂Ê≥Å„ÇíÊÉ≥ÂÆö„Åó„Ŷ„ÅÑ„Åæ„Åô„ÄÇ„Éû„ÉÉ„Éó„Å؉∏ãË®ò„Åß„Åô„Åå„ÄÅÊó¢„Å´Ôºà„Ç¢Ôºâ„ɪԺà„ǧԺâ„ÅÆÈÝòÂüü„Å´ÂàÜÈ°ûÊ∏à„Åø„Åß„ÅôÔºà„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅاßËÖ∏Ëè剪•Â§ñ„ÅÆÂÆø‰∏ª„Åß„Å؉Ωø„ÅÑ„Åæ„Åõ„Çì„ÅÆ„ÅßÔºà„ǶԺâ„ÅÆÈÝòÂüü„ÅØ„ÅÇ„Çä„Åæ„Åõ„ÇìÔºâ„ÄÇ„Åæ„Åü„ÄÅ„Ç®„ɨ„É°„É≥„Éà„ÅƉ∏ÄÈÉ®„Å´„Éä„É≥„Éê„ɺ„Ç퉪ò„Åë„Åæ„Åó„Åü„ÄÇÊï∞Â≠ó„ÅØ„Ç®„ɨ„É°„É≥„ÉàË™¨ÊòéÁÅÆÂõ≥„Å®ÂØæÂøú„Åó„Ŷ„ÅÑ„Åæ„Åô„ÄÇ„Åß„Åç„Çã„ÅÝ„ÅëËá™Âäõ„ÅßÁêÜËߣ„ÇíÊ∑±„ÇÅ„Çâ„Çå„Çã„ÅÆ„ÅØ„ÅÑ„ÅÑ„Åì„Å®„Åß„Åô„Åå„ÄʼnΩï„Åã„Çè„Åã„Çâ„Å™„ÅÑ„Åì„Å®„Å™„Å©„ÅÇ„Çå„Å∞ÂΩìÁ§æ„ÅÆË£ΩÂìÅ„Åß„Åô„ÅÆ„ÅßÂΩìÁ§æ„ÉÜ„ÇØ„Éã„Ç´„É´„ǵ„Éù„ɺ„Éà„Å´„ÅäÂïè„ÅÑÂêà„Çè„Åõ„Åè„ÅÝ„Åï„ÅÑ„ÄÇ
大腸菌の選別に使用する抗生物質は何がいいのか?
Ôºà„Ç¢Ôºâ„Å´‚ÄùAmpicillinR‚Äù„Åå„ÅÇ„Çã„ÅÆ„Åß„Ç¢„É≥„Éî„Ç∑„É™„É≥„Åå‰Ωø„Åà„Çã„Åì„Å®„Åå„Çè„Åã„Çä„Åæ„Åô„ÄÇ„Å°„Å™„Åø„Å´Ôºà„Ç¢Ôºâ„Å´„ÅÇ„Çã‚Äùrop‚Äù„ÅØoriginÔºàpBR322 oriÔºâ„ÅÆÂà∂Âæ°ÂõÝÂ≠ê„Åß„ÄÅorigin„ÅƉ∏ÄÈÉ®„ÅÝ„Å®ËÄÉ„Åà„ŶÂïèÈ°å„ÅÇ„Çä„Åæ„Åõ„Çì„ÄÇ„Åæ„Åü„ÄÅrop„ÅåÂ≠òÂú®„Åó„Ŷ„ÅфŶ„ÇÇ„Éû„ÉÉ„Éó„Å´Ë®ò˺â„Åï„Çå„Ŷ„ÅÑ„Å™„ÅфDZ„ɺ„Çπ„Åå§ö„ÅÑ„Çà„ÅÜ„Åß„Åô„ÄÇ
クローニングサイトはどこか?どのように遺伝子を挿入したらいいのか?
„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅÆ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„Å؂룄Ŵ„ÅÇ„Çä„Åæ„Åô„ÄÇÂà∂ÈôêÈÖµÁ¥Ý„ǵ„ǧ„Éà„Å؉∏¶„Çì„Åß„ÅÑ„Åæ„Åõ„Çì„Åå„ÄÅÂàá„ÇåÁõÆ„ÅåÂÖ•„Å£„Ŷ„ÅÑ„Çã„ÅÆ„Åß„Çè„Åã„Çä„ÇÑ„Åô„ÅÑ„Å®ÊÄù„ÅÑ„Åæ„ÅôÔºà‰∏ãÂõ≥ÂèÇÁÖßÔºâ„ÄÇ
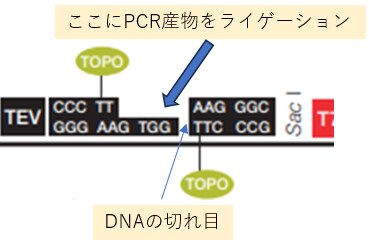
„Åì„ÅÆ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„ÅØÂΩìÁ§æÁã¨ËᙄÅÆDirectional TOPO‚Ñ¢„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„Éô„ÇØ„Çø„ɺ„Åß„Åô„ÄÇ„Åì„Çå„Çâ„ÅÆ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„ÅØ„ÄÅ„Åì„ÅƉΩçÁΩÆ„Å߉∫à„ÇÅÂàáÊñ≠„ɪÁ∑öÁä∂Âåñ„Åï„Çå„Ŷ„ÅÑ„Çã„ÅÆ„ÅßÂà∂ÈôêÈÖµÁ¥Ý„ÅßÂàá„ÇãÂøÖ˶ńÅØ„ÅÇ„Çä„Åæ„Åõ„Çì„ÄÇ„Åì„Åì„Å´PCR„Åߢó„ÇÑ„Åó„ÅüGOI„Çí„É©„ǧ„Ç≤„ɺ„Ç∑„Éß„É≥„Åó„Åæ„Åô„ÄÇ„Åì„Åì„Åß„ÅØË£ΩÂìÅ„ÅÆË©≥„Åó„ÅÑË™¨Êòé„ÅØÊéß„Åà„Åæ„Åô„Åå„ÄÅ„ÇÇ„Åó„ÅîËààÂë≥„Åå„ÅÇ„Çå„Å∞Ë£ΩÂìÅÊÉÖÂݱԺàË£ΩÂìÅÁï™Âè∑ÔºöK15101Ôºâ„Çí„ÅîÁ¢∫Ë™ç„Åè„ÅÝ„Åï„ÅÑ„ÄÇ„Åæ„Åü„ÄÅ„Ç∑„É™„ɺ„Ç∫Á¨¨1Âõû„ÅÆ„É°„É¢3„ÅßÂ∞ë„Åó„ÅÝ„ÅëË™¨Êòé„Åó„Ŷ„ÅÑ„Åæ„Åô„ÅÆ„Åß„Çà„Çç„Åó„Åë„Çå„Å∞„ÅîÂèÇÁÖß„Åè„ÅÝ„Åï„ÅÑ„ÄÇ
„Å™„Åä„ÄÅ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„Å´„Çà„Å£„Ŷ„Å؉ΩøÁÅô„ÇãPCRÈÖµÁ¥Ý„ÅÆÁ®ÆÈ°û„ÅåÈôêÂÆö„Åï„Çå„Çã„Åì„Å®„Åå„ÅÇ„Çä„Åæ„Åô„ÄÇDirectional TOPO„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„Éô„ÇØ„Çø„ɺ„ÅÆ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„ÅÆÂÝ¥Âêà„ÅØ„ÅÑ„Çè„ÇÜ„Çã„Éè„ǧ„Éï„Ç£„Éá„É™„ÉÜ„Ç£„ɺPCRÈÖµÁ¥Ý„Çí‰ΩøÁÅó„ŶPCR„ÇíË°å„ÅÜ„ÅÆ„Åå‰∏ÄËà¨ÁöÑ„Åß„Åô„ÄDŽŧ„Åæ„ÇäÊåøÂÖ•„Åô„Çã2Êú¨ÈéñDNAÊñ≠Áâá„ÅÆÊú´Á´Ø„ÅåÂπ≥ʪë„Åß„ÅÇ„Çã„Åì„Å®„ÅåÂøÖÈÝà„Åß„ÄÅ3‚ÄôAÁ™ÅÂá∫„Å剪ò„ÅèTaq DNA„Éù„É™„É°„É©„ɺ„Ǻ„ÅØÂêë„Åç„Åæ„Åõ„Çì„ÄÇ
„Çø„Ç∞ÈÖçÂàó„Å®GOI„ÅÆË™≠„ÅøÊûÝ„ÇíÂêà„Çè„Åõ„Çã„Åü„ÇÅ„Å´„ÅØ„Å©„ÅÜ„Åó„Åü„Çâ„ÅÑ„ÅÑ„ÅÆ„ÅãÔºü
„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„ÅƉ∏äʵÅÔºàT7„Éó„É≠„É¢„ɺ„Çø„ɺ„ÅƉ∏ãʵńɪÁü¢Âç∞„ÅÆÂÖà„ÅÆÊñπÔºâ„Å´RBS + ATG + 6xHis + V5 epitope + TEV„Å®„ÅÑ„ÅÜ„Ç®„ɨ„É°„É≥„Éà„ÅåÂ≠òÂú®„Åó„Åæ„Åô„ÄÇÁøªË®≥„ÅØRBSÔºà„É™„Éú„ÇΩ„Éº„ÉÝ„Éê„ǧ„É≥„Éá„Ç£„É≥„Ç∞„ǵ„ǧ„Éà„ÄÅÂà•ÂêçSDÈÖçÂàóÔºâ„ÅÆÂæå„ÅÆATG„Åã„ÇâÂßã„Åæ„Çä„Åæ„Åô„ÄÇ„Åì„ÅÆË™≠„ÅøÊûÝ„Å®GOI„ÅÆË™≠„ÅøÊûÝ„ÇíÂêà„Çè„Åõ„ÇãÂøÖ˶ńÅå„ÅÇ„Çä„Åæ„Åô„Älj∫à„ÇÅÂàá„ÇåÁõÆ„ÅåÂÖ•„Å£„Åü„Éô„ÇØ„Çø„ɺ„ÅÆÂÝ¥Âêà„ÅØ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„Å´GOI„ÅÆÂÖ®Èï∑„ÇíÂÖ•„Çå„Çå„Å∞ÂïèÈ°å„Å™„ÅÑ„Åì„Å®„Åå§ö„ÅÑ„Åß„Åô„ÄDŽŧ„Åæ„Çä„ÄÅATGÔΩû„Çπ„Éà„ÉÉ„Éó„Ç≥„Éâ„É≥„Åæ„Åß„ÅÆORFÂÖ®Èï∑„Çí„Åù„ÅÆ„Åæ„ÅæÂÖ•„Çå„Çã„Åì„Å®„Åå§ö„ÅÑ„Åß„ÅôÔºàCÊú´Á´Ø„Çø„Ç∞ÈÖçÂàó„Å®ËûçÂêà„Åï„Åõ„ÇãÂÝ¥Âêà„ÅØ„Çπ„Éà„ÉÉ„Éó„Ç≥„Éâ„É≥„ÇíÈô§ÂéªÔºâ„ÄÇ
„Åü„ÅÝ„Åó„ÄÅDirectional TOPO„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„Éô„ÇØ„Çø„ɺ„ÅÆÂÝ¥Âêà„Å؉æã§ñ„Åß„ÄÅATG„ÅÆÂâç„Å´CACC„Å®„ÅÑ„ÅÜÈÖçÂàó„Ç퉪òÂäÝ„Åô„ÇãÂøÖ˶ńÅå„ÅÇ„Çã„ÅÆ„Åß„ÅîÊ≥®ÊÑè„Åè„ÅÝ„Åï„ÅÑÔºàË©≥„Åó„Åè„ÅØË£ΩÂìÅ„Éû„Éã„É•„Ç¢„É´„Çí„ÅîÁ¢∫Ë™ç„Åè„ÅÝ„Åï„ÅÑÔºâ„ÄÇ„Å™„Åä„ÄÅGOI„ÅÆÊúÄÂàù„ÅÆATG„ÇíÊÆã„Åô„Å®„Åì„Åì„Åã„ÇâÁøªË®≥„ÅåÂßã„Åæ„ÇãÂèØËÉΩÊÄß„ÇíÂê¶ÂÆö„Åß„Åç„Å™„ÅÑ„Åü„ÇÅ„ÄÅÁâπ„Å´ÁêÜÁÅåÁÑ°„Åë„Çå„Å∞ÊúÄÂàù„ÅÆATG„ÇÇÁúÅ„ÅèÊñπ„Åå„Éô„Çø„ɺ„ÅÝ„Å®ÊÄù„ÅÑ„Åæ„Åô„ÄÇ
タンパク質発現はどのように行うのか?
„Å©„ÅÆ„Çà„ÅÜ„Å´„Çø„É≥„Éë„ÇØË≥™Áô∫Áèæ„ÇíË°å„ÅÜ„ÅÆ„Åã„Å´„ŧ„ÅфŶ„ÇÇ„Éô„ÇØ„Çø„ɺ„Éû„ÉÉ„Éó„Åã„ÇâË™≠„ÅøÂèñ„Çå„Åæ„Åô„ÄÇ„Åæ„Åö„ÄÅ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„ÅƉ∏äʵńŴT7 promoter„Åå„ÅÇ„Çä„Äʼn∏ãʵńŴT7 terminator„Åå„ÅÇ„Çã„ÅÆ„Åß„ÄÅ„ÅÑ„Çè„ÇÜ„ÇãpET„Ç∑„Çπ„ÉÜ„ÉÝ„ÇíÁÅÑ„Åü§ßËÖ∏ËèåÁô∫ÁèæÁ≥ª„Å߉ΩøÁÅß„Åç„Çã„Éô„ÇØ„Çø„ɺ„ÅÝ„Å®„ÅÑ„ÅÜ„Åì„Å®„ÅåÂàÜ„Åã„Çä„Åæ„Åô„ÄDŽŧ„Åæ„Çä„ÄÅT7 RNA„Éù„É™„É°„É©„ɺ„Ǻ„ÇíË™òÂ∞éÁô∫Áèæ„Åß„Åç„Çã„Çà„ÅÜ„Å´Êîπ§â„Åï„Çå„ÅüBL21 (DE3)„Å™„Å©„ÅƧßËÖ∏Ëèå„ÇíÁÅÑ„ÄÅIPTG„Çí‰Ωø„Å£„Ŷ˙òÂ∞éÁô∫Áèæ„Åô„Çã„Åü„ÇÅ„ÅÆ„Éô„ÇØ„Çø„ɺ„Åß„ÅÇ„Çã„Åì„Å®„Åå„Çè„Åã„Çä„Åæ„Åô„Äǂ먂Äô„Åã„ÇâÁô∫Áèæ„Åô„ÇãlacI„Çø„É≥„Éë„ÇØË≥™Ôºà„É©„ÉÉ„ÇØ„É™„Éó„ɨ„ÉɄǵ„ɺԺâ„Åå‚먄ÅÆlacO„Ç®„ɨ„É°„É≥„Éà„Å´ÁµêÂêà„Åó„ŶT7„Éó„É≠„É¢„ɺ„Çø„ɺ„Åã„Çâ„ÅÆGOI„ÅÆ˪¢ÂÜôÔºàmRNAÂêàÊàêÔºâ„ÇíÈòªÂÆ≥„Åô„Çã„Åå„ÄÅIPTG„ÇíÊ∑ªÂäÝ„Åô„Çã„Å®„É©„ÉÉ„ÇØ„É™„Éó„ɨ„ÉɄǵ„ɺ„ÅålacO„Ç®„ɨ„É°„É≥„Éà„Åã„Çâ§ñ„Çå„ŶmRNAÂêàÊàê„ÅåÈñãÂßã„Åï„Çå„Çã„Å®„ÅÑ„ÅÜ„Åó„Åè„Åø„Åß„Åô„ÄÇ
„Å™„Åä„ÄÅBL21(DE3)„Å™„Å©„ÅƧßËÖ∏ËèåÂÜÖ„Åß„ÅØT7 promoter„Åã„Çâ„ÅÆ˪¢ÂÜô„Å´ÂøÖÈÝà„ÅÆT7 RNA polymerase„ÅåÂêàÊàê„Åï„Çå„Åæ„Åô„Åå„ÄÅ„Åì„Çå„ÇÇÂêå„Åò„Åó„Åè„Åø„ÅßIPTG„Å´„Çà„ÇäË™òÂ∞é„Åï„Çå„Åæ„Åô„ÄÇ„Çà„Å£„Ŷ„ÄÅlacI„ÄÅlacO„Ç®„ɨ„É°„É≥„Éà„ÇíÊåÅ„Åü„Å™„ÅÑ„Éô„ÇØ„Çø„ɺ„ÇÇ„ÅÇ„Çä„Åæ„Åô„ÅåIPTG„Å´„Çà„ÇãÁô∫ÁèæË™òÂ∞é„ÅØ˵∑„Åì„Çä„ÄÅÊ©üËÉΩÁöфŴ§߄Åç„Å™ÂïèÈ°å„ÅØ„Å™„ÅÑ„Çà„ÅÜ„Åß„Åô„ÄÇ
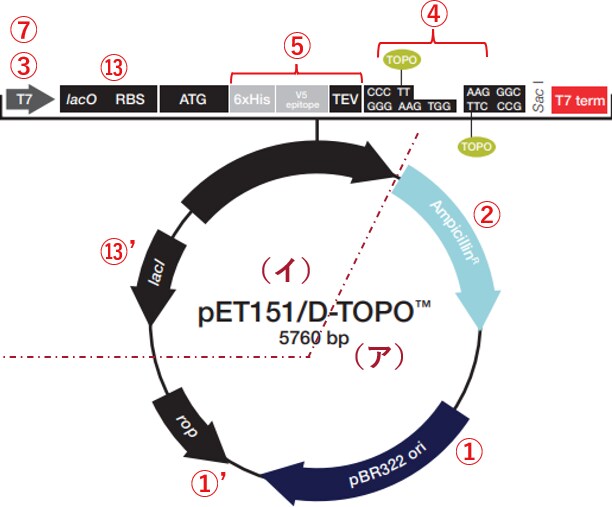
例2:一般的な大腸菌用発現ベクター(N末端タグなし、C末端タグ付き)
ÂÖàÁ®ã„Å®Âêå„ÅòpET„Ç∑„Çπ„ÉÜ„ÉÝ„ÇíÁÅÑ„Åü§ßËÖ∏ËèåÁô∫ÁèæÁ≥ªÁÉô„ÇØ„Çø„ɺ„Åß„Åô„Äǧ߄Åç„Å™ÈÅï„ÅÑ„ÅØNÊú´„Çø„Ç∞„ÅƉª£„Çè„Çä„Å´CÊú´„Çø„Ç∞Ôºà6xHisÔºâ„Ç퉪ò„Åë„Çâ„Çå„ÇãÁÇπ„Å®„ÄÅ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„Éà„ÅåÁï∞„Å™„ÇãÁÇπ„Åß„Åô„Äljæã1„Å®ÂêåÊßòÔºà„Ç¢Ôºâ„ÄÅÔºà„ǧԺâ„ÅÆ2ÈÝòÂüü„Å´ÂàÜÈ°ûÊ∏à„Åø„Åß„Åô„ÄÇÂêÑ„Ç®„ɨ„É°„É≥„Éà„Å´‰ªò„Åë„ÅüÁï™Âè∑„Å´„ŧ„ÅфŶ„ÇÇÂêåÊßò„Åß„Åô„ÄÇ
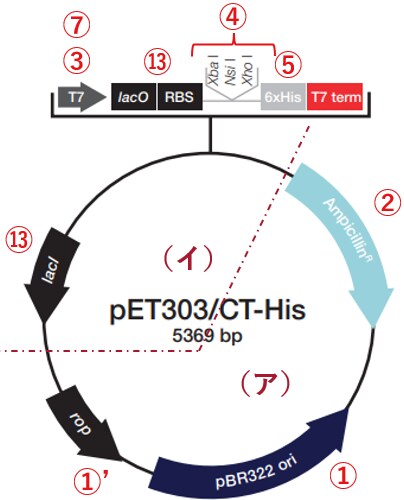
大腸菌の選別に使用する抗生物質は何がいいのか?
(ア)に”AmpicillinR”があるのでアンピシリンが使えることが分かります。
クローニングサイトはどこか?どのように遺伝子を挿入したらいいのか?
‚룄Ŵ3„ŧ„ÅÝ„Åë„Åß„Åô„ÅåÂà∂ÈôêÈÖµÁ¥Ý„ǵ„ǧ„Éà„Åå‰∏¶„Çì„Åß„ÅÑ„Åæ„ÅôÔºàXba I„ÄÅNsi I„ÄÅXho IÔºâ„ÄÇ„Åì„Åì„Å´GOI„ÇíÊåøÂÖ•„Åó„Åæ„Åô„ÄÇ„Åì„ÅƉΩçÁΩÆ„Åß„Éô„ÇØ„Çø„ɺ„ÇíÂàáÊñ≠„ɪÁ∑öÁä∂Âåñ„Åó„ÄÅDNA„É™„Ǩ„ɺ„Ǻ„Çí‰Ωø„Å£„ŶGOI„Çí„É©„ǧ„Ç≤„ɺ„Ç∑„Éß„É≥„Åô„Çã„Å®„ÅÑ„ÅÜ„ÅÆ„ÅåÊúÄ„Çlj∏ÄËà¨ÁöÑ„Å™ÊñπÊ≥ï„Åß„Åô„ÄÇ„ÅÇ„Çã„ÅÑ„ÅØGibsonÁ≠â„ÅÆ„Ç∑„ɺ„É݄ɨ„Çπ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ÅßË°å„ÅÜ„Åì„Å®„ÇÇÂèØËÉΩ„Åß„Åô„ÄÇ
„Å™„Åä„ÄÅ„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅÆÂÝ¥Âêà„ÄÅÊ≥®ÊÑè„Åô„ÇãÁÇπ„ÅØ„Åì„ÅÆÂæå„Å´Á§∫„Åô2ÁÇπ„Åß„Åô„ÄÇ„Åæ„Åü„ÄÅ„Åù„Çå„Çâ„ÇíËÄÉÊÖÆ„Åô„Çã„Å´„Å؉ª•‰∏ã„ÅÆ„Çà„ÅÜ„Å™„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„ÉàÂë®Ëæ∫„ÅÆ°©Âü∫ÈÖçÂàóÊÉÖÂݱ„ÅåÂøÖÈÝà„Åß„Åô„ÄÇ6X His„Çø„Ç∞„ÅÆË™≠„ÅøÊûÝ„Çí˵§Á∑ö„ÅßÊõ∏„ÅçË溄Åø„ÄÅÁøªË®≥ÁµÇÊ≠¢„Ç≥„Éâ„É≥ÔºàStopÔºâ„ÇíÊòéÁ§∫„Åó„Åæ„Åó„Åü„ÄÇ
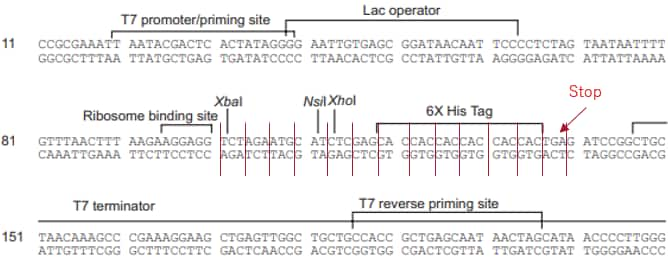
RBSからATGまでの距離が5~13塩基になるようにする
ÂäπÁéáÁöÑ„Å™ÁøªË®≥„Å´„ÅØ„ÄÅRBS„Åã„ÇâÁøªË®≥ÈñãÂßã„Ç≥„Éâ„É≥ÔºàATGÔºâ„Åæ„Åß„ÅÆË∑ùÈõ¢„ÅåÈáç˶ńÅß„Åô„ÄÇÊâÄË™¨„ÅÇ„Çä„Åæ„Åô„Åå5ÔΩû13°©Âü∫„ÅåÈÅ©Âàá„Å݄ŮˮĄÇè„Çå„Ŷ„ÅÑ„Åæ„Åô„ÄÇ„Åß„Åç„Çã„ÅÝ„ÅëË∑ùÈõ¢„Åå5ÔΩû13°©Âü∫„Å´„Å™„Çã„Çà„ÅÜ„Å´GOI„ÇíÊåøÂÖ•„Åó„Ŷ„Åè„ÅÝ„Åï„ÅÑ„ÄÇ„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅÆÂÝ¥Âêà„ÄÅXba I„ǵ„ǧ„ÉàÔºàTCTAGAÔºâ„Çí‰Ωø„ÅÑ„ÄÅGOIÈÖçÂàó„Çí„É™„É≥„Ç´„ɺÁÑ°„Åó„ÅßÁõ¥Áµê„Åô„Çã„Å®RBS„Å®ATG„ÅÆË∑ùÈõ¢„ÅØ6°©Âü∫ÔºàÈñì„Å´6°©Âü∫ÂÖ•„ÇãÔºâ„Åß„Åô„ÄÇ„Å™„Åä„ÄÅNsi I„ǵ„ǧ„ÉàÔºàATGCATÔºâ„ÇíÂà©ÁÅï„Çå„ÇãÂÝ¥Âêà„ÅØÂ∞ë„ÅóÁâπÊÆä„Å™Ê≥®ÊÑè„ÅåÂøÖ˶ńÅß„Åô„ÄÇNsi I„ǵ„ǧ„Éà‰∏≠„Å´ATG„Åå„ÅÇ„Çä„ÄÅÁøªË®≥ÈñãÂßã„Ç≥„Éâ„É≥„Å®„Åó„ŶʩüËÉΩ„Åô„Çã„Åü„ÇÅ„Åß„Åô„ÄÇNsi I„ǵ„ǧ„Éà„ÅƉ∏ãʵńŴGOIÈÖçÂàó„ÄÅ„ÅÇ„Çã„ÅÑ„ÅØGOIÈÖçÂàó„Åã„ÇâATG„ÇíÈô§Â骄Åó„ÅüÈÖçÂàó„ÇíÁõ¥Ê镄ŧ„Å™„Åê„Åì„Å®„Å´„Å™„Çä„Åæ„Åô„Åå„ÄÅNsi I„ǵ„ǧ„Éà‰∏≠„ÅÆATG„Åã„ÇâÁøªË®≥„ÅåÂßã„Åæ„Çã„Åü„ÇÅ„ÄÅATGCAT„ÅåÁøªË®≥„Åï„Çå„Åü„É°„ÉÅ„Ç™„Éã„É≥„ÄÄ„Éí„Çπ„ÉÅ„Ç∏„É≥„ÅåÈÝ≠„Å´„ŧ„Åç„Åæ„Åô„ÄÇ„Åì„ÅÆÂÝ¥Âêà„ÇÇRBS„Å®ATG„ÅÆÈñì„ÅÆ°©Âü∫Êï∞„ÅØ6„Åß„Åô„Åå„ÄÅ„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅÆÂÝ¥ÂêàÂÜÖÈÉ®ÈÖçÂàó„Å´Xba I„ǵ„ǧ„Éà„Åå„Å™„Åë„Çå„Å∞Xba I„ǵ„ǧ„Éà„ÇíÂà©ÁÅó„ÅüÊñπ„Åå„Çà„Åï„Åù„ÅÜ„Åß„Åô„ÄÇ
CÊú´„Çø„Ç∞Ôºà6xHisÔºâ„Ů˙≠„ÅøÊûÝ„ÇíÂêà„Çè„Åõ„Çã„ɪ„Çπ„Éà„ÉÉ„Éó„Ç≥„Éâ„É≥„ÇíÈô§Â骄Åô„Çã
‰æã„Åà„Å∞Xba I„ǵ„ǧ„Éà„Å®Xho I„ǵ„ǧ„ÉàÔºàCTCGAGÔºâ„ÅÆÈñì„Å´GOI„ÇíÊåøÂÖ•„Åô„ÇãÂÝ¥Âêà„Å´„ŧ„ÅфŶ„Åß„Åô„ÄÇXho IÈÖçÂàó„ÇíCÊú´Á´Ø„ÅÆ6xHis„Çø„Ç∞„Å´Âêà„Çè„Åõ„ÅüË™≠„ÅøÊûÝ„ÅßÂå∫Âàá„Çã„Å®CTC GAG„Åß„Åô„ÅÆ„Åß„ÄÅ„Çπ„Éà„ÉÉ„Éó„Ç≥„Éâ„É≥„ÇíÈô§Â骄Åó„ÅüGOI„Çí„É™„É≥„Ç´„ɺÁÑ°„Åó„ÅßXho I„ǵ„ǧ„Éà„Å´Áõ¥Áµê„Åô„Çã„Å®CÊú´„Çø„Ç∞Ôºà6xHis„Çø„Ç∞Ôºâ„Ů˙≠„ÅøÊûÝ„ÅåÂêà„ÅÑ„Åæ„Åô„ÄÇ„ÇÇ„ÅóCÊú´„Çø„Ç∞„ÇíËûçÂêà„Åï„Åõ„Åü„Åè„Å™„ÅÑÂÝ¥Âêà„ÅØÈÅ∫‰ºùÂ≠ê„ÅÆ„Çπ„Éà„ÉÉ„Éó„Ç≥„Éâ„É≥„ÇíÊÆã„Åó„Ŷ„Åè„ÅÝ„Åï„ÅÑ„ÄÇ„Å™„Åä„ÄÅ2„ÅãÊâÄ„ÅßÂàáÊñ≠„Åô„ÇãÂÝ¥Âêà„Äʼn∏°„ǵ„ǧ„Éà„ÅÆË∑ùÈõ¢„ÅåÊúĉΩé2°©Âü∫Á®ãÈõ¢„Çå„Ŷ„ÅÑ„ÇãÂøÖ˶ńÅå„ÅÇ„Çä„Åæ„Åô„ÄÇ„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅÆÂÝ¥Âêà„ÅØXba I„Å®Xho I„ÅÆË∑ùÈõ¢„Åå6°©Âü∫‰ª•‰∏äÊÖ£„Çå„Ŷ„ÅÑ„Çã„ÅÆ„ÅßÂïèÈ°å„ÅÇ„Çä„Åæ„Åõ„Çì„ÄÇ
タンパク質発現はどのように行うのか?
例1と同じpET系大腸菌発現用ベクターですのでタンパク質発現方法は全く同じです。
例3:昆虫細胞発現で使うバキュロウイルス作成用ベクター(Bac-to-Bac Baculovirus Expression System用)
ÊòÜËô´Á¥∞ËÉûÁô∫Áèæ„Å´‰ΩøÁÅô„ÇãÁµÑ„ÅøÊèõ„Åà„Éê„Ç≠„É•„É≠„Ƕ„ǧ„É´„Çπ‰ΩúÊàêÁÉô„ÇØ„Çø„ɺ„Åß„Åô„ÄÇÁµÑ„ÅøÊèõ„Åà„Éê„Ç≠„É•„É≠„Ƕ„ǧ„É´„Çπ„Çí‰ΩúÊàê„Åô„ÇãÊñπÊ≥ï„ÅØ˧áÊï∞ÈñãÁô∫„Åï„Çå„Ŷ„ÅÑ„Åæ„Åô„Åå„ÄÅ„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅØÂΩìÁ§æ„ÅÆGibco‚Ñ¢ Bac-to-Bac‚Ñ¢ Baculovirus Expression SystemÁÅß„Åô„ÄÇÊòÜËô´Á¥∞ËÉû„ÅßÊ©üËÉΩ„Åô„Çã„Éù„É™„Éò„Éâ„É™„É≥„Éó„É≠„É¢„ɺ„Çø„ɺԺàpPHÔºâ„Å®„Éê„Ç≠„É•„É≠„Ƕ„ǧ„É´„Çπ„Ç≤„Éé„ÉÝÔºàBacmidÔºâ„Å®„ÅÆÁµÑ„ÅøÊèõ„Åà„ǵ„ǧ„ÉàÔºàTn7R„ÄÅTn7LÔºâ„Åå„ÅÇ„Çã„Åì„Å®„ÅåÁâπÂ楄Åß„Åô„ÄÇ„Å™„Åä„ÄÅÔºà„Ç¢Ôºâ„ŮԺà„ǶԺâ„Åå‰∫å„ÅãÊâÄ„Åö„ŧ„ÅÇ„Çä˧áÈõë„Åß„Åô„Åå„ÄÅ3ÈÝòÂüü„Å´ÂàÜÈ°ûÊ∏à„Åø„Åß„Åô„ÄÇÁï™Âè∑„ÅØÂÖàÁ®ã„ÅƉæã„Å®ÂêåÊßò„Åß„Åô„ÄÇ
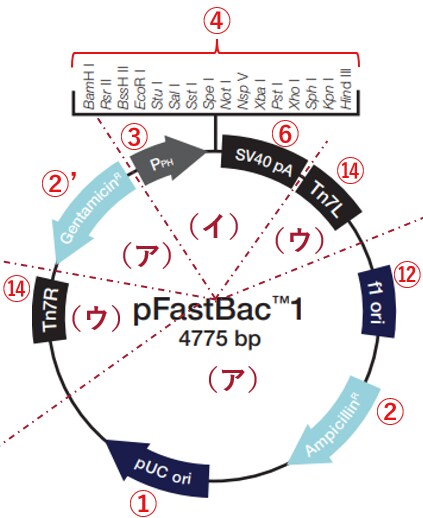
大腸菌の選別に使用する抗生物質は何がいいのか?
(ア)に”AmpicillinR”があるのでアンピシリンが使えることが分かります。なお、実はゲンタマイシンも使用可能です。②’のゲンタマイシン耐性遺伝子(GentamicinR)の上流にプロモーターの記載がなく分かりにくいですが、何も記載がなければ大腸菌用のプロモーターが省略されている可能性が非常に高く、このベクターも然りです。
クローニングサイトはどこか?どのように遺伝子を挿入したらいいのか?
‚룄Ŵ„Éû„É´„ÉÅ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„ÉàÔºàMCSÔºâ„Åå„ÅÇ„Çä„Åæ„Åô„ÅÆ„Åß„ÄÅ„Åì„Åì„Å´Âà∂ÈôêÈÖµÁ¥Ý„Å®DNA„É™„Ǩ„ɺ„Ǻ„ÇíÁÅÑ„ÅüÊñπÊ≥ï„ÄÅ„ÅÇ„Çã„ÅÑ„ÅØGibsonÁ≠â„ÅÆ„Ç∑„ɺ„É݄ɨ„Çπ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞Ê≥ï„ÅßGOI„ÇíÊåøÂÖ•ÂèØËÉΩ„Åß„Åô„ÄÇ„Éô„ÇØ„Çø„ɺ„Å´ËûçÂêà„Çø„Ç∞ÈÖçÂàó„ÅØ„ÅÇ„Çä„Åæ„Åõ„Çì„ÅÆ„Åß„ÄÅÁâπ„Å´Ë™≠„ÅøÊûÝ„ÇíÂêà„Çè„Åõ„ÇãÂ∑•Â§´„ÅØÂøÖ˶ńÅÇ„Çä„Åæ„Åõ„Çì„ÄÇMCS„ÅÆ„Å©„Åì„Åß„ÇÇ„ÅÑ„ÅÑ„Çè„Åë„Åß„Åô„Åå„Äʼnª•‰∏ã„ÅÆÂü∫Ê∫ñ„ÅßÊåøÂÖ•ÂÝ¥ÊâÄ„ÇíÈÅ∏„Å∞„Çå„Çã„ÅÆ„Åå„ÅäÂãß„ÇÅ„Åß„Åô„ÄÇ
- GOI„Å´Â≠òÂú®„Åó„Å™„ÅÑÂà∂ÈôêÈÖµÁ¥Ý„ÇíÈÅ∏ÊäûÔºàDNA„É™„Ǩ„ɺ„Ǻ„Çí‰ΩøÁÅô„ÇãÂÝ¥ÂêàÔºâ
- „É©„Éú„Å´„ÅÇ„Çã„ɪÂÆâ‰æ°„Å™„ɪ§ö„Åè„ÅÆ„É°„ɺ„Ç´„ɺ„Åã„ÇâË≤©Â£≤„Åï„Çå„Ŷ„ÅÑ„ÇãÂà∂ÈôêÈÖµÁ¥Ý„ÇíÈÅ∏Êäû
- „Åß„Åç„Çå„Å∞MCS„ÅƉ∏äʵńى∏ãʵńÅƉ∫å„Ç´ÊâÄÔºà„Åß„Åç„Çã„ÅÝ„Åë‰ΩôÂàÜ„Å™ÈÖçÂàó„ÇíÊÆã„Åï„Å™„ÅÑÊñπ„Åå‰ΩôË®à„Å™ÂèØËÉΩÊÄß„ÇíËÄÉ„Åà„ÇãÂøÖ˶ńÅå„Å™„ÅÑ„ÅÆ„ÅßÂÆâÂøÉ„Å®„ÅÑ„ÅÜÁ®ãÂ∫¶„ÅÆÁêÜÁî±Ôºâ
タンパク質発現はどのように行うのか?
当社のBac-to-Bac™ Baculovirus Expression System用ベクターですので以下の流れです。
- pFastBac1のような専用ベクターにGOIを組み込む
- Â∞ÇÁî®Â§ßËÖ∏ËèåÔºàDH10BacÔºâ„Å´‰∏äË®ò„ÇíÂ∞éÂÖ•„Åó„ÄŧßËÖ∏ËèåÂÜÖÈÉ®„ÅßGOI„ÇíÁµÑ„ÅøË溄Çì„ÅÝÁµÑ„ÅøÊèõ„ÅàBacmid„ÇíË™øË£Ω„Åô„Çã
- 精製組み換えBamidを昆虫細胞へトランスフェクションで導入し、組み換えバキュロウイルスを作成
„Åï„Çâ„Å´Ë©≥„Åó„Åè„ÅäÁü•„Çä„Å´„Å™„Çä„Åü„ÅÑÊñπ„ÅØË£ΩÂìÅ„Éû„Éã„É•„Ç¢„É´ÔºàË£ΩÂìÅÁï™Âè∑Ôºö10359016Ôºâ„Çí„ÅîÁ¢∫Ë™ç„ÅÑ„Åü„ÅÝ„Åè„Åã„ÄÅÂΩìÁ§æ„ÉÜ„ÇØ„Éã„Ç´„É´„ǵ„Éù„ɺ„Éà„Å´„ÅäÂïèÂêà„Åõ„Åè„ÅÝ„Åï„ÅÑ„ÄÇ
例4:酵母Pichia発現用ベクター
ʨ°„ÅØPichia pastoris„Å®„ÅÑ„ÅÜÈÖµÊØç„ÇíÁÅÑ„ÅüÁô∫ÁèæÁ≥ªÁÉô„ÇØ„Çø„ɺ„ÅƉæã„Åß„Åô„ÄÇPichia pastoris„ÅØÁúüÊÝ∏ÁîüÁâ©„Åß„Åô„ÅåÊØî˺ÉÁöÑÂÆâ‰æ°„Åß„Åã„ŧȴòÁô∫Áèæ„ÇíÊúüÂæÖ„Åß„Åç„Çã„Åü„ÇÅ„ÄŧßËÖ∏ËèåÁô∫ÁèæÁ≥ª„ÅßÂèØÊ∫∂ÊÄßÁô∫Áèæ„ÅåÂè∂„Çè„Å™„Åã„Å£„ÅüÂÝ¥Âêà„Å™„Å©„Å´Ë©¶„Åï„Çå„Çã„Åì„Å®„Åå§ö„ÅÑ„Åß„Åô„ÄÇ„Åæ„Åü„ÄŧßËÖ∏Ëèå„Å®ÈÅï„ÅÑÂàÜÊ≥åÁô∫Áèæ„ÅåÂæóÊÑè„Åß„Äʼnæã„Åà„Å∞„ǵ„ǧ„Éà„Ç´„ǧ„É≥„Å™„Å©„ÅƧßÈáèË™øË£Ω„ÅßÂÆüÁ∏æ„Åå„ÅÇ„Çä„Åæ„Åô„ÄÇ„Å°„Å™„Åø„Å´„Åì„ÅÆ„Éô„ÇØ„Çø„ɺ„ÅØÂàÜÊ≥åÁô∫ÁèæÁÅß„Åô„ÄÇ„Å™„Åä„ÄÅ‚ëÝÔΩû‚뢄ÅÆÈÝòÂüü„Å´ÂàÜÈ°ûÊ∏à„Åø„Åß„Åô„ÄÇ„Çè„Åã„Çä„Å´„Åè„ÅÑ„Åß„Åô„Åå5‚Äô AOX1„ÅåGOIÁô∫ÁèæÁÅÆ„Éó„É≠„É¢„ɺ„Çø„ɺ„Åß„ÄÅ„Åù„ÅƉ∏ãʵńÅÆŒ±-factor„ÅåÂàÜÊ≥å„Ç∑„Ç∞„Éä„É´„Åß„Åô„ÄÇÁï™Âè∑„ÅØ„Ç®„ɨ„É°„É≥„ÉàË™¨Êòé„ÅÆÂõ≥„Å®ÂØæÂøú„Åó„Ŷ„ÅÑ„Åæ„Åô„ÄÇ
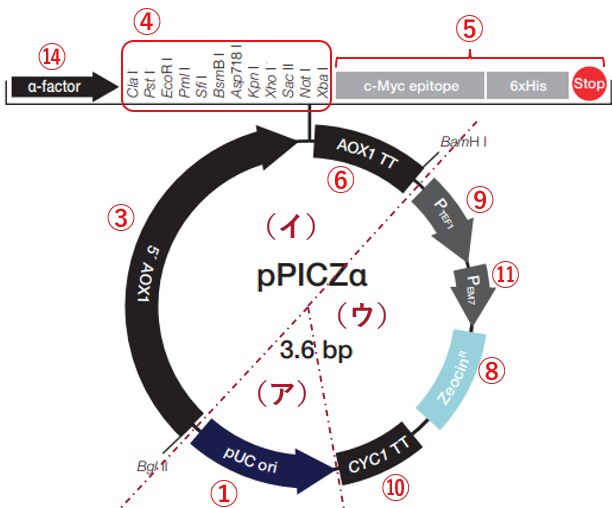
Ôºà„Ç¢Ôºâ„Å´ÊäóÁîüÁâ©Ë≥™ËÄêÊÄßÈÅ∫‰ºùÂ≠ê„Åå˶ãÂΩì„Åü„Çä„Åæ„Åõ„Çì„ÄÇ„Åù„ÅÆÂÝ¥Âêà„ÅØÁѶ„Çâ„Åö„Å´Ôºà„ǶԺâ„Çí˶ã„Ŷ„Åè„ÅÝ„Åï„ÅÑ„ÄÇÈÖµÊØçÁÅÆËÄêÊÄßÈÅ∫‰ºùÂ≠êZeocinR„ÅÆÔºàÁü¢Âç∞„ÅÆÈÝ≠„ÅåÁúÅÁï•Ôºü„Åï„Çå„Ŷ„ÅфŶ„Çè„Åã„Çä„Å´„Åè„ÅÑ„Åß„Åô„ÅåÔºâ‰∏äʵńŴ§ßËÖ∏ËèåÁÅÆEM7„Éó„É≠„É¢„ɺ„Çø„ɺ„Åå„ÅÇ„Çã„Åì„Å®„Åã„Çâ„ÄŧßËÖ∏Ëèå„ÅÆÈÅ∏Âà•„Å´„ÇÇZeocin„Çí‰ΩøÁÅß„Åç„Çã„Åì„Å®„Åå„Çè„Åã„Çä„Åæ„Åô„ÄÇÊú¨„Éô„ÇØ„Çø„ɺ„ÅÆ„Çà„ÅÜ„Å´„ÄÅ„Éô„ÇØ„Çø„ɺ„ǵ„ǧ„Ç∫„ÇíÂ∞è„Åï„Åè„Åô„Çã„Åü„ÇńŴ§ßËÖ∏Ëè剪•Â§ñ„ÅÆ„Éõ„Çπ„ÉàÁî®ÈÅ∏Êäû„Éû„ɺ„Ç´„ɺ„٧ßËÖ∏ËèåÁî®ÈÅ∏Êäû„Éû„ɺ„Ç´„ɺ„ÇíÂÖ±ÁÅó„Ŷ„ÅÑ„Çã„DZ„ɺ„Çπ„ÅØ„Åó„Å∞„Åó„Å∞˶ãÂèó„Åë„Çâ„Çå„Åæ„Åô„ÄÇ
クローニングサイトはどこか?どのように遺伝子を挿入したらいいのか?
‚룄Ŵ„Éû„É´„ÉÅ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ǵ„ǧ„ÉàÔºàMCSÔºâ„Åå„ÅÇ„Çä„Åæ„Åô„ÅÆ„Åß„ÄÅ„Åì„Åì„Å´Âà∂ÈôêÈÖµÁ¥Ý„Å®DNA„É™„Ǩ„ɺ„Ǻ„ÇíÁÅÑ„ÅüÊñπÊ≥ï„ÄÅ„ÅÇ„Çã„ÅÑ„ÅØGibsonÁ≠â„Ç∑„ɺ„É݄ɨ„Çπ„ÇØ„É≠„ɺ„Éã„É≥„Ç∞Ê≥ï„ÅßGOI„ÇíÊåøÂÖ•ÂèØËÉΩ„Åß„Åô„ÄÇÊ≥®ÊÑè„ÅåÂøÖ˶ńř„ÅÆ„ÅØGOI„ÅƉ∏°ÂÅ¥„ÅßË™≠„ÅøÊûÝ„ÇíÂêà„Çè„Åõ„ÇãÂøÖ˶ńÅå„ÅÇ„ÇãÁÇπ„Åß„Åô„ÄDŽŧ„Åæ„Çä„Äʼnª•‰∏ã„ÅÆ„Çà„ÅÜ„Å™Âõ≥„ÇíÂèÇËÄÉ„Å´„Åó„Ŷ„ÄÅNÊú´ÂÅ¥„ÅÆÂàÜÊ≥åÁÇ∑„Ç∞„Éä„É´„Éö„Éó„ÉÅ„ÉâÈÖçÂàó(α-factor signal sequenceÔΩûSte13 signal cleavageÈÖçÂàó„Åæ„ÅßÔºâ„Å®CÊú´ÂÅ¥„ÅÆËûçÂêà„Çø„Ç∞Ôºàc-myc epitope + polyhistidine tagÔºâ„ÅƉ∏°Êñπ„Å®GOI„ÅÆË™≠„ÅøÊûÝ„ÇíÂêà„Çè„Åõ„ÇãÂøÖ˶ńÅå„ÅÇ„Çä„Åæ„Åô„ÄÇ„ÇÇ„Å°„Çç„ÇìCÊú´Á´Ø„Çø„Ç∞„ÇíËûçÂêà„Åï„Åõ„Å™„ÅÑÂÝ¥Âêà„ÅØGOI„ÅÆ„Çπ„Éà„ÉÉ„Éó„Ç≥„Éâ„É≥„ÇíÊÆã„Åó„ÄÅ„Åã„ŧ˙≠„ÅøÊûÝ„ÅØÊ∞ó„Å´„Åô„ÇãÂøÖ˶ńÅØ„ÅÇ„Çä„Åæ„Åõ„Çì„ÄÇÂõ≥„Å´„ÅØNÊú´ÂÅ¥„ÅÆË™≠„ÅøÊûÝ„Çí˵§Á∑ö„ÄÅCÊú´ÂÅ¥„ÅÆË™≠„ÅøÊûÝ„ÇíÈùíÁ∑ö„ÅßÁ§∫„Åó„Åæ„Åó„Åü„ÄÇÁøªË®≥„ÅØŒ±-factor signal sequence„ÅÆÈÝ≠„ÅÆATG„Åã„ÇâÂßã„Åæ„Çä„Åæ„Åô„ÄÇ
※クリックして画像を拡大
„Å™„Åä„ÄÅ„Å©„ÅÜ„Åó„Ŷ„ÇÇGOI„ÅÆNÊú´Á´Ø„Å´‰ΩôË®à„Å™ÈÖçÂàó„Ç퉪ò„Åë„Åü„Åè„Å™„ÅÑÂÝ¥Âêà„Å´„Å®„ÇãÊñπÊ≥ï„Å´„ŧ„ÅфŶ‰ª•‰∏ã„Å´Á∞°Âçò„Å´Ë™¨Êòé„Åó„Åæ„Åô„ÄÇ
‰æã„Åà„Å∞EcoR I„ǵ„ǧ„ÉàÔºàGAATTCÔºâ„Å´GOI„Çí„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„Åô„ÇãÂÝ¥Âêà„ÅØGAA TTC GOIÈÖçÂàó„Å®„Å™„Çä„ÄÅGOIÈÖçÂàó„ÅÆNÊú´„Å´Glu Phe„ÅÆ2„Ç¢„Éü„ÉéÈÖ∏„Å剪òÂäÝ„Åï„Çå„Åæ„Åô„ÄÇ„Åß„Åç„Çã„ÅÝ„ÅëGOI„ÅÆNÊú´„Å´‰Ωï„Çljªò„Åë„Åü„Åè„Å™„ÅÑÂÝ¥Âêà„ÅÆ„ÅäÂãß„ÇÅ„ÅÆÊñπÊ≥ï„Å؉ª•‰∏ã„ÅÆÈÄö„Çä„Åß„Åô„ÄÇ
- ベクターをKex2 signal cleavageの少し上流のXho Iサイトで切断(MCSにも一つXho Iサイトがある点要注意)
- GOI„ÅÆ5‚ÄôÊú´„Å´PCR„ÅßXho I„Åã„ÇâKex2 signal cleavage„ǵ„ǧ„ÉàÔºàAAA AGA„Åæ„ÅßÔºâ„Ç퉪òÂäÝ
- 上記をXho Iで切断したものをベクターにライゲーション
上記の方法で行うと、Kex2 singal cleavageの直後にGOIが挿入され、Kex2によるプロセシング(切断)がうまくいけば、GOIのN末端に余計な配列が付かないことになります。
タンパク質発現はどのように行うのか?
„Éû„ÉÉ„Éó„Åã„Çâ„Å©„ÅÆ„Çà„ÅÜ„Å´ÊⱄÅÜ„Åã„ÇíË™≠„ÅøÂèñ„Çå„Åæ„Åô„Åå„ÄÅÂÖ®„ÅèPichiaÁô∫ÁèæÁ≥ª„Å´„ŧ„ÅфŶ„ÅîÂ≠ò„Åò„Å™„ÅÑÂÝ¥Âêà„ÅØ„ÅäÊâãÊï∞„Åß„Åô„Åå„Éû„Éã„É•„Ç¢„É´„Çí„ÅîÁ¢∫Ë™ç„Åè„ÅÝ„Åï„ÅÑ„ÄÇ„Åæ„Åü„Äʼn∏çÊòéÁÇπ„ÅÇ„Çå„Å∞ÂΩìÁ§æ„ÉÜ„ÇØ„Éã„Ç´„É´„ǵ„Éù„ɺ„Éà„Çí„ÅîÂà©ÁÅè„ÅÝ„Åï„ÅÑ„ÄÇ
以下に簡単に流れを説明します。
- ベクターのMCSにGOIをクローニング
- 5‚ÄôAOX1„ÅƉ∏Ä„ÅãÊâÄ„ÅßÂà∂ÈôêÈÖµÁ¥ÝÂàáÊñ≠‚ÜíÁ∞°ÊòìÁ≤æË£Ω
- Pichia pastorisにエレクトロポレーション等で導入
- Zocinで選別
- タンパク質発現(メタノールで誘導)
Pichia„Ç≤„Éé„ÉÝ„ÅÆAOX1„ǵ„ǧ„ÉàÔºà„É°„Çø„Éé„ɺ„É´Ë≥áÂåñ„Å´Èñ¢„Çè„ÇãÈÖµÁ¥Ý„ÅÆ„Éó„É≠„É¢„ɺ„Çø„ɺÈÝòÂüüÔºâ„Å´Áõ∏ÂêåÁµÑ„ÅøÊèõ„Åà„Åß„Éô„ÇØ„Çø„ɺ‰∏∏„Åî„Å®„ÇíÊåøÂÖ•„Åó„Åæ„Åô„ÄÇ„Éô„ÇØ„Çø„ɺ„ÇíÂàá„Çâ„Å™„Åè„Ŷ„ÇÇÂÖ•„Çã„ÅØ„Åö„Åß„Åô„Åå„ÄÅÂäπÁéá„Çí‰∏ä„Åí„Çã„Åü„ÇÅ„Å®„Äʼn∫àÊúü„Åõ„ŨÂÝ¥ÊâÄ„Å´ÂÖ•„ÇãÁ¢∫Áéá„Çí‰∏ã„Åí„Çã„Åü„ÇÅ„Å´„Äʼn∫à„ÇÅ„Éô„ÇØ„Çø„ɺ„ÅÆAOX1ÈÖçÂàó‰∏ä„Åß„Ç´„ÉÉ„Éà„Åô„Çã„Åì„Å®„Çí„ÅäÂãß„ÇÅ„Åó„Ŷ„ÅÑ„Åæ„Åô„ÄÇÂΩ¢Ë≥™Ëª¢ÊèõÂæåZeocin„ÅßÂΩ¢Ë≥™Ëª¢ÊèõÊÝ™„ÇíÈÅ∏Âà•„Åó„Åæ„Åô„ÄÇ
ÊúÄÂæå„Å´
2回にわたって、マップからベクターの使い方を読み取る方法をご説明しました。少しでもご参考になったでしょうか?
„Éû„ÉÉ„Éó„Çí˶ã„Çã„Å®„ÄÅÊâãÂÖÉ„ÅÆ„Éô„ÇØ„Çø„ɺ„Åå„Å©„Çì„Å™„ÇÇ„ÅÆ„Åã„ÅÝ„Åë„Åß„Å™„Åè„ÄÅÊⱄÅÑÊñπ„ÇÑÂÆüÈ®ì„ÅÆÈÄ≤„ÇÅÊñπ„ÇÇË™≠„ÅøÂèñ„Çå„Åæ„Åô„ÄÇˮĄÅÑÊèõ„Åà„Çã„Å®„ÄÅ„Éû„ÉÉ„Éó„ÅØ„Éô„ÇØ„Çø„ɺ„ÇíÁÅÑ„ÅüÂÆüÈ®ì„ÅÆ„Éû„Éã„É•„Ç¢„É´„Åß„ÄÅ„Éû„ÉÉ„Éó„Åã„ÇâÂÆüÈ®ì„ÅÆÈÄ≤„ÇÅÊñπ„Åæ„ÅßË™≠„ÅøÂèñ„ÇãÂøÖ˶ńÅå„ÅÇ„Çã„Å®„ÇÇˮĄÅà„Åæ„Åô„ÄÇ„Åó„Åã„Åó„ÄÅË™≠„ÅøÊûÝ„ÇíÂêà„Çè„Åõ„ÇãÂÝ¥Âêà„Å™„Å©„Éû„ÉÉ„É󉪕§ñ„ÅÆÊÉÖÂݱ„ÅåÂøÖ˶ńŴ„Å™„Çã„Åì„Å®„ÇÇ„ÅîÁêÜËߣ„ÅÑ„Åü„ÅÝ„Åë„Åü„Åã„Å®ÊÄù„ÅÑ„Åæ„Åô„ÄÇ„Åì„ÅÆ4Âõû„ÅÆ„Éñ„É≠„Ç∞„ÇíÈÄö„Åó„Ŷ„ÄÅ„Éû„ÉÉ„Éó„Åã„Çâ„Éô„ÇØ„Çø„ɺ„ÇíÊⱄÅ܉∏ä„Åß„ÅÆÊúĉΩéÈôê„ÅÆÊÉÖÂݱ„ÇíË™≠„ÅøÂèñ„Çå„Çã„Çà„ÅÜ„Å´„Å™„Å£„Ŷ„ÅÑ„Åü„ÅÝ„Åç„ÄÅ„Åù„ÅƉªñÂøÖ˶ńřÊÉÖÂݱ„ÇíË£ΩÂìÅ„Éû„Éã„É•„Ç¢„É´„Å´Ê预Åó„Å´Ë°å„Åë„Çã„Çà„ÅÜ„Å´„Å™„Çå„Å∞„Å®ÊÄù„ÅÑ„Åæ„Åô„ÄÇ„Åæ„Åü„ÄÅ„É°„ɺ„Ç´„ɺ„ÅÆ„ÉÜ„ÇØ„Éã„Ç´„É´„ǵ„Éù„ɺ„Éà„Çlj∏äÊâã„Å´Ê¥ªÁÅó„Ŷ„Åè„ÅÝ„Åï„ÅÑ„ÄÇ
„Åæ„Åü„ÄÅ„ÅîÂà©ÁÅÑ„Åü„ÅÝ„ÅèÁô∫ÁèæÁ≥ª„Å´„ŧ„ÅфŶ„ÅîÁêÜËߣ„ÅÑ„Åü„ÅÝ„Åè„Åì„Å®„ÅØ„Éô„ÇØ„Çø„ɺ„Éû„ÉÉ„Éó„Çí„Çà„ÇäË©≥„Åó„Åè„ɪ„Çπ„Éû„ɺ„Éà„Å´ÁêÜËߣ„Åô„Çã„Åü„ÇÅ„ÅÆÊó©ÈÅì„Åß„Åô„ÄÇ„Åù„ÅÆ„ÅÇ„Åü„Çä„Å´„ŧ„ÅфŶ„Åæ„ÅüÂà•„ÅÆ„Éñ„É≠„Ç∞„Åß„ÅäË©±„Åß„Çå„Å∞„Å®ÊÄù„ÅÑ„Åæ„Åô„ÄÇ
„ÅîÊÑè˶ãÁ≠â„Åî„Åñ„ÅÑ„Åæ„Åó„Åü„ÇâÊú´Â∞æ„ÅÆ„Éï„Ç©„ɺ„ÉÝ„Åß„ÅäÂݱ„Åõ„Åè„ÅÝ„Åï„ÅÑ„ÄÇ
ÂÆüÈöõ„ÅäÊåÅ„Å°„ÅÆ„Éó„É©„Çπ„Éü„Éâ„Éô„ÇØ„Çø„ɺ„ÅÆËߣ˙≠„ǵ„Éù„ɺ„Éà„ÅÑ„Åü„Åó„Åæ„ÅôԺńÇÇ„Å°„Çç„ÇìÂΩìÁ§æ„ÅÆ„ÇÇ„ÅÆ„Åß„Å™„Åè„Ŷ„ÇÇÊßã„ÅÑ„Åæ„Åõ„Çì„ÄÇ„Éû„ÉÉ„Éó„Çí„ÉÜ„ÇØ„Éã„Ç´„É´„ǵ„Éù„ɺ„ÉàÂÆõ„ŶԺàjptech@thermofisher.comÔºâ„Å´„ÅäÈÄÅ„Çä„ÅÑ„Åü„ÅÝ„Åè„Åã„ÄÅÊú¨Ë®ò‰∫ã„ÅƉ∏ÄÁ∏ã„Å´„ÅÇ„Çã„Éï„Ç©„ɺ„ÉÝ„Åã„Çâ„Åî‰æùÈݺ„Åè„ÅÝ„Åï„ÅÑ„ÄÇ
テクニカルセミナー(無料)
ベクターマップの読み方~クローニング・タンパク質発現で迷子にならないために
お申込みはこちら
ハンズオントレーニング(有償)
初めてのクローニング ハンズオントレーニング
お申込みはこちら
ベクターマップの読み方ガイド 無料ダウンロード
【無料公開中】Protein Expressionハンドブック
„Çø„É≥„Éë„ÇØÁô∫Áèæ„ÅÆÂÆüÈ®ì„ÇíʧúË®é„Åó„Ŷ„ÅÑ„ÇãÊñπ„ÇÑ„ÄÅË©≥Á¥∞ÊÉÖÂݱ„ÇíÁ¢∫Ë™ç„Åó„Åü„ÅÑÊñπ„Å´„ÅÜ„Å£„Ŷ„ŧ„Åë„ÅÆ„Éè„É≥„Éâ„Éñ„ÉÉ„ÇØ„Åß„Åô„ÄÇ
ダウンロードはこちら
本ブログに関連するリンク
- „Éô„ÇØ„Çø„ɺ„Éû„ÉÉ„Éó„ÅÆË™≠„ÅøÊñπÔΩû„ÇØ„É≠„ɺ„Éã„É≥„Ç∞„ɪ„Çø„É≥„Éë„ÇØË≥™Áô∫Áèæ„ÅßËø∑Â≠ê„Å´„Å™„Çâ„Å™„ÅÑ„Åü„ÇÅ„Å´„ÄÄ Á¨¨1Âõû„ÄÄ„Éó„É©„Çπ„Éü„Éâ„Éô„ÇØ„Çø„ɺ„ÅÆÊßãÈÄÝ„Å®„Éû„ÉÉ„Éó„Åã„ÇâÂàÜ„Åã„Çã„Åì„Å®
- ベクターマップの読み方~クローニング・タンパク質発現で迷子にならないために  第2回 プラスミドベクターに含まれるエレメントリスト
- ベクターマップの読み方~クローニング・タンパク質発現で迷子にならないために  第3回 ベクターマップの読み方・実例集1
Zeocin is a trademark of InvivoGen.
ÁÝîÁ©∂ÁŴ„ÅÆ„Åø‰ΩøÁÅß„Åç„Åæ„Åô„ÄÇË®∫Êñ≠ÁŴ„Å؉ΩøÁÅÑ„Åü„ÅÝ„Åë„Åæ„Åõ„Çì„ÄÇ




