▼もくじ
はじめに
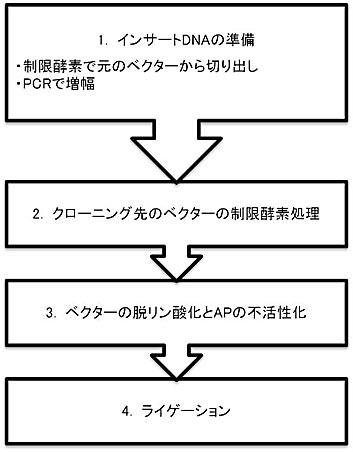
クローニングした遺伝子の機能を解析するために、サブクローニング(あるプラスミドの断片を、別のプラスミドに組み込むこと)は分子生物学研究で必須な技術です。一般的な実験の流れは、インサートDNAの準備、クローニング先のベクターの制限酵素処理、ベクターの脱リン酸化とalkaline phosphatase (AP)の不活性化、ライゲーションとなります(図1)。制限酵素の種類やライゲーションに用いる試薬などによっては、これらの工程は1日以上要することもあります。
このプロセスは、分子生物学実験を行う際にはほぼ必須ではありますが、他の重要な実験の時間を奪ってしまっているのも事実ではないでしょうか?
そこで今回は、この工程を最短で1.5時間に短縮できる弊社の製品を用いたFast Cloningの方法を紹介します。
▼こんな方におすすめです!
・クローニング実験に多くの時間を要している方
・クローニング実験をよりすばやく行いたい方
インサートDNAの準備
サブクローニングするためには、まず制限酵素やPCRを用いてインサートDNAの準備を行います。
制限酵素処理で元のベクターからインサートDNAを準備する場合
一般的には2種類の制限酵素で切断します。従来の制限酵素を用いた二重切断の場合、制限酵素の組み合わせで反応バッファーを替える必要があり、完全な切断には数時間から一昼夜かかります。また、長時間の反応ではスター活性*を気にしなくてはいけません。
(スター活性*:制限酵素の特異性が低下して、本来のターゲットとは異なる塩基配列を切断してしまう現象のこと。)
一方で、FastDigest Enzymesは、176種類のすべての酵素が1つのFastDigest reaction buffer(またはFastDigest Green reaction buffer)で100%の活性を持つため、バッファーを替えることなく二重切断できます。また、切断速度が速く5-15分でDNAを切断できるため、スター活性を気にしなくて済みます(図2)。さらにFastDigest Green Bufferを用いた反応液を使えば、電気泳動のゲルに直接ローディングすることも可能です。

図2 FastDigest enzymesで5分間処理したプラスミドDNAの単および二重切断。M: GeneRuler 1 kb Plus DNA Ladder、C: 未切断プラスミドDNA、1: FastDigest SmaIで5分間切断したプラスミドDNA、2: FastDigest EcoRIで5分間切断したプラスミドDNA、3: FastDigest SmaIとFastDigest EcoRIで5分間二重切断したプラスミドDNA。
インサートDNAをPCRで準備する場合
正確性の高いPCR酵素を用います。
例えば、Pfu polymeraseを用いた場合、正確性はTaq polymeraseと比べて8倍です。一方、この酵素では1.5 kbの増幅断片を伸長するのに1時間要します。Phusion High-Fidelity DNA PolymeraseはPyrococcus由来の新規プルーフリーディング酵素に2本鎖DNA結合ドメインを融合させたPCR酵素です。2本鎖DNA結合ドメインは、DNA polymeraseとDNAの間の結合を物理的に安定化させ、1回の結合あたりに取り込めるヌクレオチドの量を増やします。また、伸長反応に必要な結合イベントの回数を減らします。この技術によって、Taqと比べて52倍の正確性(Phusion HF Bufferにおけるエラー率は4.4 x 10-7)で、1.5 kbの増幅断片の増幅が最短20分で完了します(図3)。
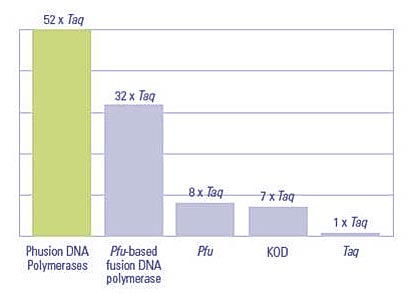
図3 異なるPCR酵素の正確性の比較。Phusion DNA Polymeraseのエラー率はTaq DNA polymeraseより52倍、Pfu DNA polymeraseより6.5倍低くなりました。
クローニング先のベクターの制限酵素処理
準備したインサートDNAを目的のベクターにクローニングするためには、適切な制限酵素によってベクターを切断します。先に記した通り、FastDigest enzymesを使用することでDNAの切断が5-15分で可能です。
ベクターの脱リン酸化とAPの不活性化
制限酵素処理後はセルフライゲーション*を防ぐために脱リン酸化処理をしますが、Calf Intestinal Alkaline Phosphatase (CIAP)を用いた場合、脱リン酸化とアルカリホスファターゼの不活化反応の時間を入れると約1.5時間要します。一方、FastAP Thermosensitive Alkaline Phosphataseを使うと、37℃・10分で迅速に脱リン酸化し、75℃・5分の高温処理により不活化します(図4)。また、FastDigest reaction bufferおよびFastDigest Green reaction buffer中で活性を持つため、制限酵素処理したDNAの反応液に直接加えることができます。この時、制限酵素を不活化するための高温処理は必要ありません。
(*セルフライゲーション:切断したベクターDNA自身がライゲーションすること。)

図4 75°CインキュベーションによるFastAP Thermosensitive Alkaline Phosphataseの不活化。 DNA脱リン酸化反応溶液(200 units/ml phosphatase)は37°Cで30分間インキュベーションしました。その後、75℃で不活化の処理を行い、グラフに示した時間ごとにp-NPPアッセイによって残存するphosphatase活性を測定しました。
ライゲーション
インサートDNAとベクターを連結するにはリガーゼによるライゲーション反応を行います。T4 DNA Ligaseを用いた場合、ライゲーション反応には1時間から一昼夜要します。それに対し、Rapid DNA Ligation Kitは、T4 DNA ligaseを用いて1時間反応したときと同等のライゲーション反応効率を室温5分間のインキュベーションで実現します(図5)。加えて、ライゲーション反応液は、TransformAid Bacterial Transformation Kitまたは他の手法によって作製した大腸菌のコンピテントセルを用いた形質転換に直接使用できます。

図5 Rapid DNA Ligation Kitによる突出末端と平滑末端のライゲーション。 突出末端のライゲーションはLambda DNAの2.1 kb PstI断片をPstIで切断後に脱リン酸化処理したpUC19 DNA の中に挿入することで行いました。平滑末端のライゲーションはLambda DNA の2.3 kb PvuII断片をSmaIで切断後に脱リン酸化処理したpUC19の中に挿入することで行いました。ライゲーション効率は大腸菌XL1-Blue株の形質転換によって決定しました。
まとめ
1.5 kbのインサートDNAをPCRで準備した場合、従来のStandard Cloning製品とFast cloning製品を用いたPCR増幅からライゲーション反応までの時間を比較すると、前者では最短でも5.5時間要しますが、後者では最短1.5時間に短縮できます(表1)。
| Standard Cloningワークフロー | Fast Cloningワークフロー | |
| 1.5 kb DNA断片の増幅 | Pfu Polymerase Fidelity 8x Taq 1時間 |
Phusion DNA Polymerase Fidelity 52x Taq 20分 |
| 電気泳動 | 1時間 | GeneRuler DNA ladders 45分 |
| 制限酵素処理 | 1-16時間 | FastDigest enzymes 5-15分 |
| ベクターの脱リン酸化とAPの不活性化 | CIAP 1.5時間 |
FastAP Alkaline Phosphatase 15分 |
| ライゲーション | T4 DNA Ligase 1-16時間 |
Rapid DNA Ligation Kit 5分 |
| 合計 | 5.5 時間以上 | 1.5 時間 |
ライゲーション後のステップはこちら
大腸菌の形質転換について
詳しくはこちらの記事をご覧ください。
目的の遺伝子を持った組換え体をスクリーニングする方法について
詳しくはこちらの記事をご覧ください。
これだけは知っておきたいクローニングの基礎知識、ココにあります!
Invitrogen 分子生物学教室では、分子生物学の教育のメインとして、初心者だけでなく経験のある分子生物学者のために、豊富で信頼できる技術的な内容をご提供しています。基礎知識を学ぶ時にはいつでも、Invitrogen 分子生物学教室の無料PDFを活用してください!
研究用にのみ使用できます。診断用には使用いただけません。