高純度で分解のないRNAを調製することは、遺伝子発現解析を行う上で、基本的で重要なポイントです。
遺伝子発現解析には、RT-PCR、リアルタイムRT-PCR、次世代シーケンス解析、マイクロアレイ解析、ヌクレアーゼプロテクションアッセイ、RNAマッピング、in vitro翻訳、cDNAライブラリーの作成など、実にさまざまなアプリケーションが挙げられます。いずれのアプリケーションにおいてもスタートサンプルとなるRNAのクオリティが実験成功の重要なファクターとなっています。
クオリティの高いRNAの調製には、適切なRNA精製キットの選択はもちろんのこと、サンプルの保存方法やラボ環境なども十分に配慮する必要があります。
このブログでは以下の点に注意しながら、RNA精製のためのポイントをまとめています:
・サンプルの適切な保存方法
・RNA実験に適したラボ環境
・RNA精製が難しいサンプル/扱いやすいサンプル
・サンプル/アプリケーションに適したRNA精製キットの選択
▼もくじ [非表示]
RNA解析用のサンプルの適切な保存方法
サンプルの採取からRNAの精製までは迅速に行う必要があります。
一度生体から摘出された組織内のRNAは不安定で、細胞内の内在性RNaseによって分解が進みます。また細胞へのストレスなどの影響で一過的に発現が誘導される遺伝子も存在します。こうした細胞内の発現変動は遺伝子によって異なるため、予測が難しいです。
このことから正確な遺伝子発現解析を行うためには、採取後直後の遺伝子発現プロファイルを維持した状態でRNA精製に進むことが重要なポイントになります。RNAの分解を抑えた適切な組織/細胞の処理法/保存法としてサンプリング後、直ちに以下の①~③の処理をお薦めしています:
①直ちにRNA精製操作へ進む
②液体窒素で瞬間的に凍結し、-80℃のディープフリーザーで保存する
③Invitrogen™ RNAlater™ Stabilization Solution中に浸漬した状態で保存する
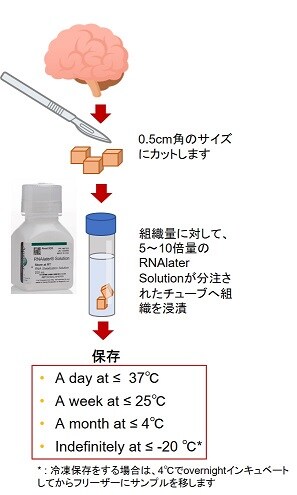
図1 RNAlater™ Stabilization Solution 操作プロトコール
サンプリング後、直ちにRNA精製へ進むことがもっとも理想的ですが、複数のサンプルをまとめて処理する場合や、サンプリングする場所と精製する場所が離れている場合は、サンプルを一定時間放置することになり、直ちに精製へ進むことは難しくなります。この間にもRNAの分解や発現変動は進んでしまいますので、サンプルが放置される可能性がある場合は、以下の保存方法を検討する必要があります。
凍結保存は、液体窒素による瞬間凍結後、-80℃で保存します。凍結保存法はRNA解析用のサンプル保存として有効です。ただ一旦、融解させてしまうと細胞内のRNAは大幅に分解が進んでしまうため、凍結したサンプルはRNA精製のホモジナイズ処理をスタートする直前まで凍結状態を厳密にキープする必要があります。凍結したサンプルを安全に融解させる試薬として、Invitrogen™ RNAlater™-ICE Frozen Tissue Transition Solutionを活用できます。RNAlater Stabilization Solutionは、操作がシンプル(図1)で、そのRNAの安定化効果も高いため(図2)、すでに多くの研究者から信頼され実績のある組織保存液です。直ちにRNA精製ができない場合や、遠隔地からのサンプルの輸送など、さまざまなシーンで利用されています。RNAlater Stabilization Solutionは、試薬成分が組織中の細胞内へ短時間で浸潤することにより、細胞内外の存在するRNase活性およびその他の活性酵素を強く抑制し、細胞内のRNAの発現プロファイルを維持させます。ほとんどの組織サンプルに利用可能な試薬ですが、試薬の浸潤性の悪いことが予想される組織(骨、脂肪など)や、RNase活性が著しく高い組織(膵臓など)には適しませんので、こうしたサンプルを保存する場合は、凍結保存法の方が適しています。
 図2 RNAlater™ Stabilization Solution 処理した組織サンプルから精製したtotal RNAの安定性の確認 マウスから摘出した組織サンプルを5倍量のRNAlater Solutionに浸漬しました。サンプルは37℃で1日間、室温(25℃)で1週間、4℃で1か月保存した後、RNAを精製し、電気泳動後(上図)、Norhtern Blotで P53、GAPDHの検出を行いました(下図)。RNAlater Solution中で保存した組織内のtotal RNAの完全性を示しています。
図2 RNAlater™ Stabilization Solution 処理した組織サンプルから精製したtotal RNAの安定性の確認 マウスから摘出した組織サンプルを5倍量のRNAlater Solutionに浸漬しました。サンプルは37℃で1日間、室温(25℃)で1週間、4℃で1か月保存した後、RNAを精製し、電気泳動後(上図)、Norhtern Blotで P53、GAPDHの検出を行いました(下図)。RNAlater Solution中で保存した組織内のtotal RNAの完全性を示しています。
RNA実験に適したラボ環境を確認
 図3 RNaseZap®を用いたRNaseの除染方法
図3 RNaseZap®を用いたRNaseの除染方法
RNAを扱う場合、ラボの環境にも注意を払う必要があります。
RNAを分解するRNaseは、空気中やラボのデスクなどの表面などあらゆる場所に存在します。水や試薬などにわずかに存在する微生物もRNaseコンタミの原因となりますし、実験操作を行う研究者の肌や体液(唾や汗、涙、粘液など)にもRNaseは含まれます。
特にラボの中でプラスミドベクターの調製を行っている場合はラボ自体がRNaseにコンタミしている可能性がありますので特に注意が必要です。プラスミドベクターの精製の際に大量に添加されるRNase Aや、クローニングに使用する酵素(制限酵素など)がRNaseコンタミの原因になるためです。
RNaseはラボ内に常に存在すると想定し、ラボの環境を管理することが必要です。定期的なRNase汚染除去の処理を行い、RNA実験用の試薬を特別に準備/管理し、RNase-freeのラボ環境を整えてください。サーモフィッシャーサイエンティフィックはRNAを扱うラボ環境の整備のためにさまざまな製品を取りそろえています。
RNase汚染除去用試薬ではInvitrogen™ RNaseZap™ RNase Decontamination Solutionなどが有効です。使用方法はシンプルで、除染したい器具の表面にRNaseZapを処理した後、RNase-free waterでリンスをして、乾かすのみの操作でRNAの実験環境を整えることができます(図3)。RNaseのコンタミネーションが疑われる器具などを短時間で除染できます。またRNA実験専用のラボでも1週間に1回程度は除染処理を行うことが理想的です。
RNaseの除染試薬の他、RNase-freeの水、試薬、チップやチューブなどの消耗品もRNA実験専用に準備し、しっかりと管理することが重要です»
RNA精製が難しいサンプル/扱いやすいサンプル
RNA精製を行う上で、取り扱うサンプルの性質を理解することは実験を進める上で重要です。
組織でもRNase活性が非常に高いものや、比較的RNase活性が低いものが存在します(図4)。RNase活性が高い組織はサンプリング後の短時間でRNAの分解が進んでしまうため、RNA精製キットのLysis buffer中で迅速に組織のホモジナイズを行い、短時間で完全に溶解させることが重要です。RNA精製キットのLysis bufferには高濃度のグアニジン塩やフェノールなどの有機溶媒が含まれているため、Lysis buffer中にRNAが溶出された状態まで進むと、RNAは比較的安定になるので、RNase活性の高い組織ほど迅速なホモジナイズ処理が必要になります。
 図4 組織別のRNase活性レベルの違い 組織により細胞内のRNase活性レベルが異なります。RNase活性が低い脳組織内のRNase活性を “1”としたときの各組織のRNase活性のレベルをまとめています
図4 組織別のRNase活性レベルの違い 組織により細胞内のRNase活性レベルが異なります。RNase活性が低い脳組織内のRNase活性を “1”としたときの各組織のRNase活性のレベルをまとめています
組織の硬固さのレベルを把握することも重要です(図5)。RNA精製では、精製キットのLysis bufferに完全に溶解しないとRNA収量が低下します。ただ硬固な組織ではホモジナイズに時間が掛かってしまう傾向があり、この間にRNAの分解が進んでしまう場合もあります。そのため硬固な組織を使用する場合は、より破砕能力の高いホモジナイズ方法を選択する必要があります。やわらかい組織にはペッスルなどを用いて手動で処理することもありますが、硬固な組織ではローター・スターター式のホモジナイザーやミキサーミルなどの機器を組み合わせる必要があります。こうした機器がない場合、液体窒素中で組織を乳鉢/乳棒で粉砕してからLysis bufferに添加してホモジナイズを行うことも有効です。
 図5 組織別のホモジュナイズの難しさ 組織によりその硬固のレベルが異なります。骨組織や心臓や骨格筋組織のような繊維組織ではホモジュナイズが難しく時間が掛かるため、分解が進んでしまったり、収量も低くなる傾向があります。
図5 組織別のホモジュナイズの難しさ 組織によりその硬固のレベルが異なります。骨組織や心臓や骨格筋組織のような繊維組織ではホモジュナイズが難しく時間が掛かるため、分解が進んでしまったり、収量も低くなる傾向があります。
なお、RNA発現レベルは組織によって異なります。そのためスタートサンプルの組織重量を揃えても、組織の種類によりその収量は大きく異なります(図6)。つまり、用いるサンプルからどの程度のtotal RNAが回収されるかを正しく予想を立てて実験計画を立てることも重要です。
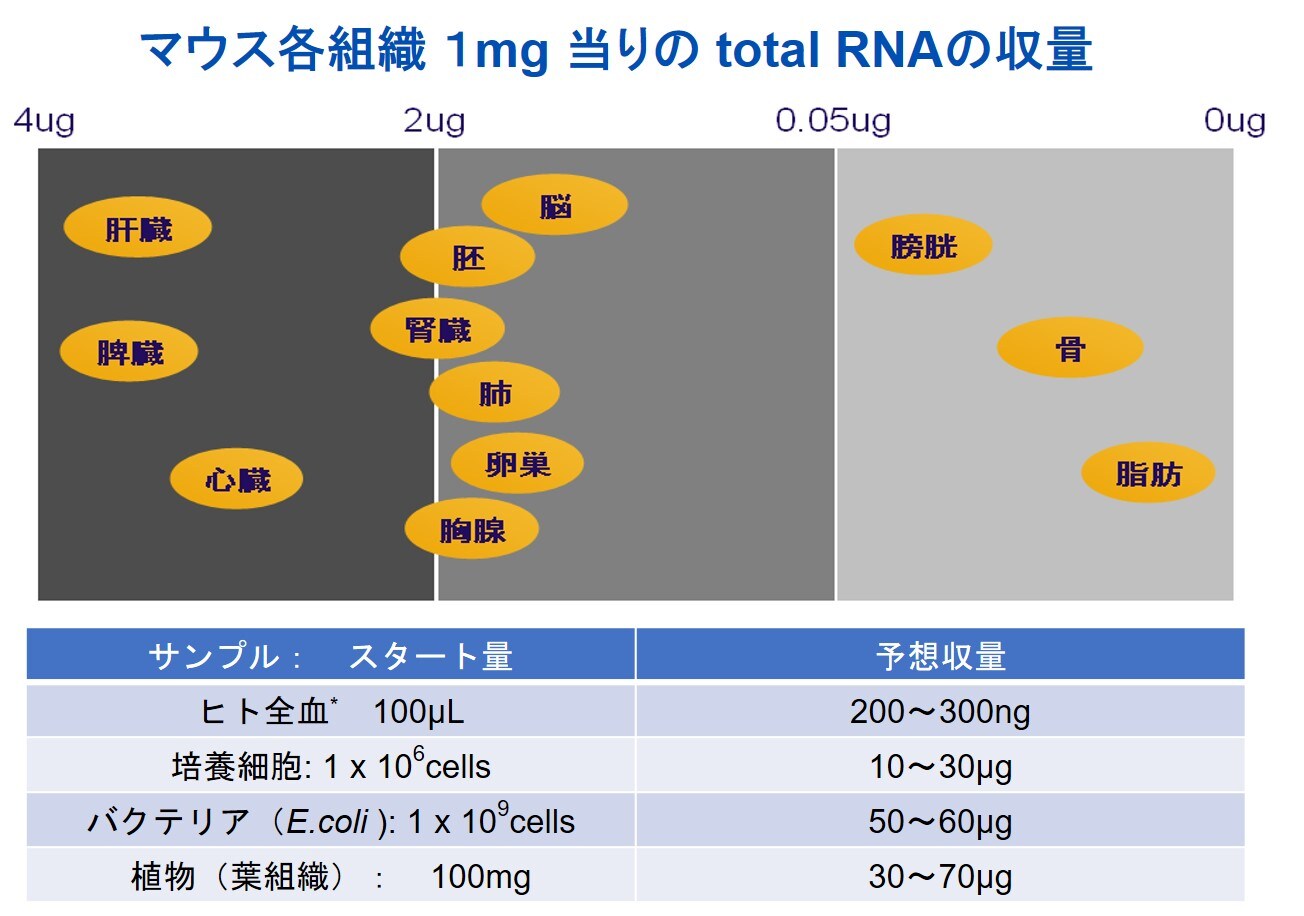 図6 組織別の予想収量 組織により細胞内のRNAの発現レベルが異なるため、同じ1mg の組織片からスタートしても回収されるtotal RNA量は異なります
図6 組織別の予想収量 組織により細胞内のRNAの発現レベルが異なるため、同じ1mg の組織片からスタートしても回収されるtotal RNA量は異なります
サンプル/アプリケーションに適したRNA精製キットの選択
表1:RNA精製システムの特徴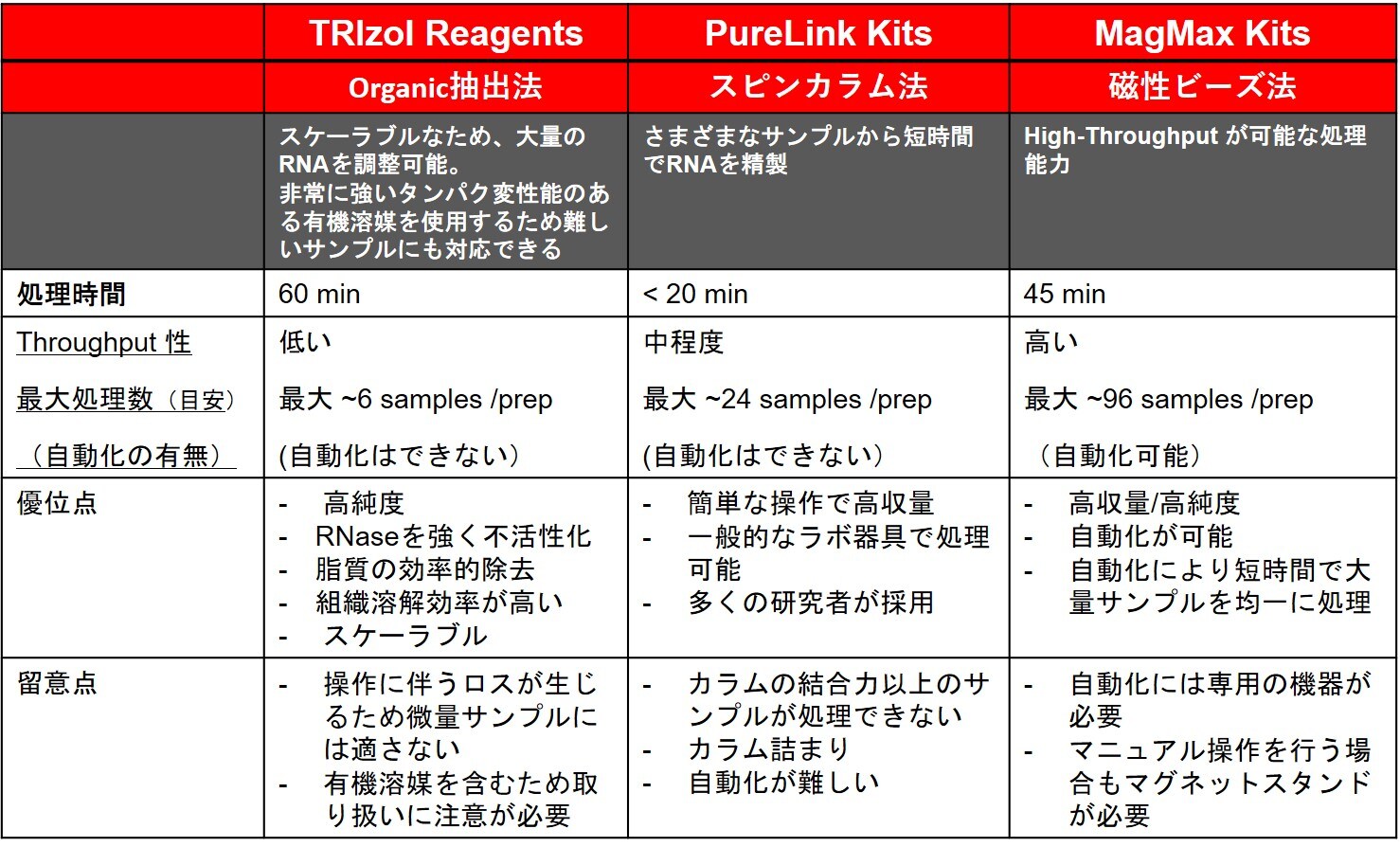
使用するサンプルに適したRNA精製キットを選択するには、まず精製システムの特長を把握する必要があります。RNA精製システムは、大きくOrganic抽出法、スピンカラム法、磁性ビーズの3種類に分けられます(表1)。
Organic抽出法として、RNA精製にはAGPC法(Acid_Guanidinium thiocyanate_Phenol _Chloroform)をベースとした抽出試薬が使用されています。古くから使用されているRNA精製システムです。AGPC Lysis bufferは非常に強力なタンパク変性能を有するため、RNase活性が非常に高いサンプルやホモジナイズが困難なサンプルからでも効率よく高純度なRNAが精製でき、現在も多くの研究者に利用されています。さらに脂肪組織などに含有する大量の脂質も?有機溶媒により効果的に除去できるうえ、AGPC Lysis bufferのpHのコントロールによってDNAの排除もできるため、スピンカラム法では難しいとされているサンプルからも純度の高いRNAが回収できます。
AGPC法ではスピンカラム法より高純度なRNAが回収できますが、試薬中のフェノールのコンタミネーションが問題となる場合があります。そのためAGPC Lysis bufferによるライセート調製とスピンカラム精製を組み合わせる手法が取られる場合もあります。サーモフィッシャーサイエンティフィックでは、AGPC法試薬としてInvitrogen™ TRIzol™ Reagentシリーズを提供しており、スピンカラムと組み合わせたTRIzol™ Plus RNA Purification Kitシリーズを提供しています。詳細はこちらをご覧ください。
スピンカラム法は、短時間で簡単に高純度なRNAを精製するシステムとして広く利用されています。シリカメンブレンのスピンカラムを用いた方法で、フェノールなどの有機溶媒を使用せずに短時間(約20分)で高純度のRNAを精製します。操作工程でのロスが少ないため、微量サンプルからのRNA精製に適しています。
スピンカラム法をベースとした精製キットは、スタートのサンプルの種類によってLysis条件を変えているものが多いため、キットの選択に注意してください。特にmiRNAなどのsmall RNAを研究する場合は、small RNAが回収できるキットであるかを確認してください。当社は、Invitrogen™ PureLink™ RNAシリーズを始めとした多くのキットを提供しています。サンプルにもっとも適したキットを選択頂けます。詳細はこちらをご覧ください。
磁性ビーズ法はスピンカラム法で採用されているシリカを磁性ビーズ表面にコーティングし、同様な結合原理でビーズ表面にDNAを結合するようにデザインされています。RNAが結合した状態で磁性ビーズはマグネットに集積することができるため、洗浄bufferや溶出bufferの交換/回収は遠心操作を行わずに処理できます。
またスピンカラムでは遠心操作の際にサンプルがメンブレンを通過する一瞬の間にRNAを結合/洗浄しますが、磁性ビーズ法ではRNAとビーズは一定時間buffer中でミックスされながらインキュベートされるため、回収効率/精製効率ともにカラムより優れています。
また磁性ビーズ法の特徴として自動化システムに適している点も挙げられます。Applied Biosystems™ MagMAX™シリーズのDNA精製システムのほとんどは、自動化機器のThermo Scientific™ KingFisher™機器(図7)に最適化済みで、Ready-to-useのプログラムがKitおよびサンプルタイプごとに作成済みになっており、簡単な設定で大量のサンプルが処理できます。詳細はこちらをご覧ください。
 図7 MagMaxシリーズに適した自動精製機器 KingFihserシリーズ
図7 MagMaxシリーズに適した自動精製機器 KingFihserシリーズ
スタートサンプルの難易度、処理サンプル数(スループット)などの条件から、Organic抽出法、スピンカラム法、磁性ビーズのいずれかを選択した後、さらにスタートサンプルの質に合わせて適する精製キットを選択してください(図8)
 図8 サンプル別 RNA精製kitの選択ガイド
図8 サンプル別 RNA精製kitの選択ガイド
まとめ
高純度で分解のないRNAを精製するためには、RNAを十分に理解し、サンプリングから保存、ラボ環境、精製方法を確認し、十分な準備と計画を立てて実験に臨む必要があります。
RNA精製操作で気をつけなくていけないポイント(トップ10)はこちらから確認できます。
[無料公開中]DNA/RNAリファレンスガイドPDF版
本リファレンスガイド(英語)は核酸精製に関するツール、ワークフロー、アプリケーションがわかるハンドブックです。みなさまの研究活動にぜひご活用ください。
研究用にのみ使用できます。診断用には使用いただけません。


