▼もくじ [非表示]
はじめに
RNA干渉(RNAi)は、広範囲な細胞タイプにおけるタンパク質機能を解析するために遺伝子発現をノックダウンする手法で、細胞の基礎生物学を研究するための非常に強力なツールです。以前は限定された研究室のみで使用されていたRNAi 技術ですが、現在では遺伝子機能研究に欠かせないものとなっています。本技術は、タンパク質ノックダウン研究、表現型解析、機能回復、パスウェイ解析、in vivoノックダウン、および創薬ターゲット探索のための卓越したツールになっています。
今回は、RNAiとノンコーディングRNAについてまとめましたのでご紹介します。
RNAiの一般用語集
RNAi
リボ核酸干渉(1998年にA. Fire and C. Melloらが初めて実施)。
siRNA
短い干渉RNA。siRNAはジヌクレオチド3’オーバーハングが付いた21–25 bpのdsRNAで、RNA干渉経路において長鎖dsRNAがDicerにより切断されたものです。合成siRNAを哺乳類細胞に導入することにより、RNAiが誘導されます。また、siRNAには内在性前駆体由来のものもあります。
shRNA
短いヘアピンRNA(短い干渉ヘアピン)。shRNAは、安定した遺伝子サイレンシングに用いられるsiRNAを供給するためのベクターベースのアプローチに用いられます。強力なPol III系プロモーターは、細胞内siRNA機構において、様々な長さのヘアピン型およびループ型のRNAを形成するようにデザインされたターゲット配列の転写を開始させるために用いられます。shRNAが細胞内に導入されると、RNAiによって相補配列の遺伝子発現が減少します。
miR RNAi
RNAiを引き起こすためのmicroRNAを発現させるためのベクター。miRNAは、19~23 ntの一本鎖RNAで、不完全な塩基対で形成されるヘアピン構造を特徴とする一本鎖の前駆体転写物から生じます。miRNAは、RISCと同一ではありませんが類似したサイレンシング複合体中で機能します(図1参照)。
化学的改変siRNA
化学的に改変されたsiRNA分子。
RISC
RNA誘導サイレンシング複合体(RISC)。タンパク質とsiRNAから構成されるヌクレアーゼ複合体で、RISC複合体内に取り込まれたsiRNAは相補的な内在性mRNAを標的として切断します。
オフターゲット効果
特定のターゲット遺伝子以外の1個または2~3個の遺伝子に生じる効果で、siRNAまたはd-siRNAプールの導入後に遺伝子機能の抑制がみられます。この効果は、siRNAのセンス鎖によって媒介され、意図しない遺伝子の機能喪失を開始させます。標的とは無関係な遺伝子の発現がノックダウンされるのに十分な相同性がある場合は、オフターゲット効果が特定のsiRNAのアンチセンス鎖の二次的効果として生じることもあります。
RNAiの作用機序
RNAiには、2タイプのsmall RNA分子が用いられます。1つは、合成された短い干渉RNA(siRNA)分子で、mRNAの切断をターゲットとして、目的遺伝子の発現を効率的にノックダウンします。これに対し、microRNA(miRNA)分子は、自然発生する19~22ヌクレオチド長の一本鎖RNAで、標的mRNAの3’非翻訳領域(UTR)に結合し、翻訳を阻害することによって、遺伝子発現を調節します(Ambros,2004)。
siRNA解析
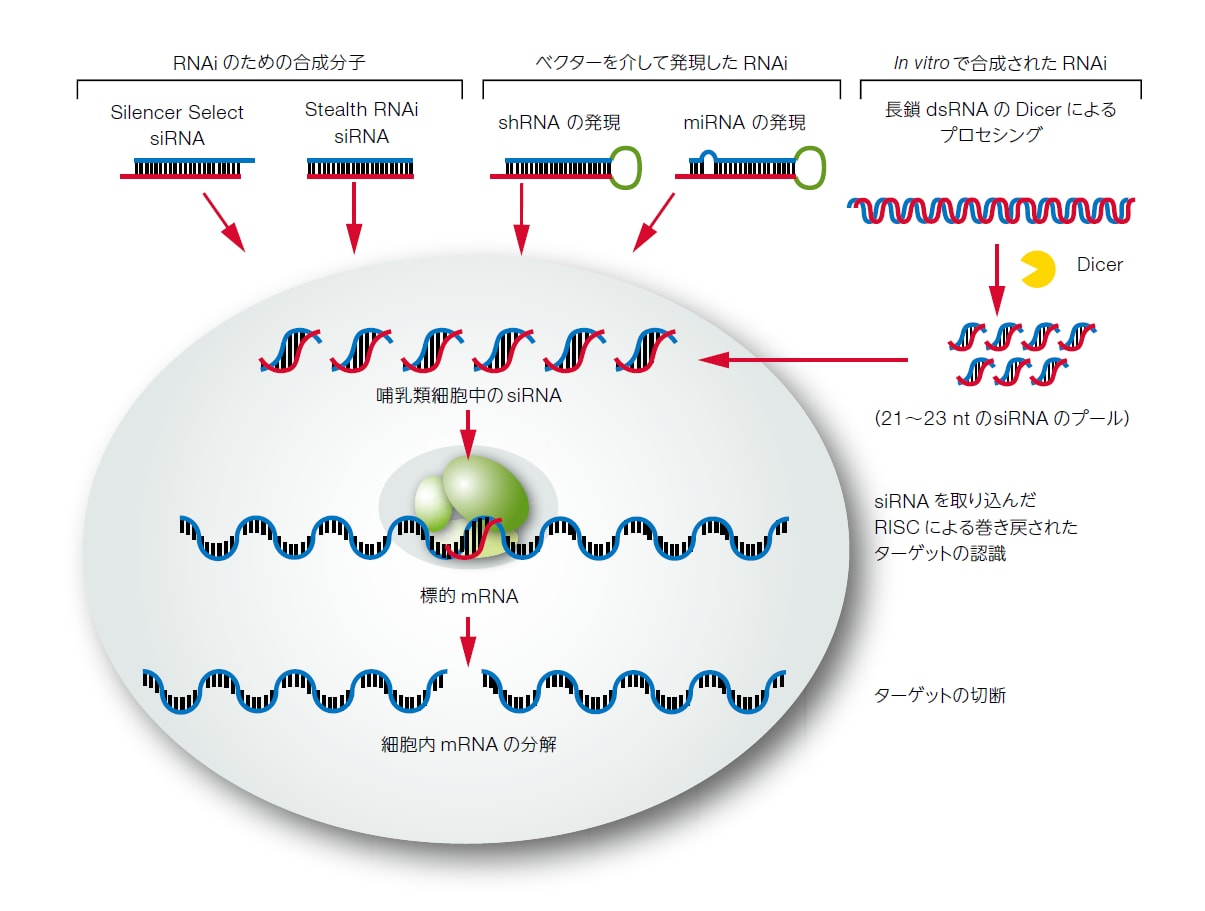
RNAiを誘導するためには、合成分子、RNAiベクター、およびin vitro dicingを用いる複数の方法があります(図1参照)。哺乳類細胞では、dsRNAの短い断片–短い干渉RNA–がターゲット細胞内mRNAの特異的分解を開始します。このプロセスにおいて、siRNAのアンチセンス鎖は多タンパク質複合体またはRNA誘導サイレンシング複合体(RISC)の一部となり、その後、対応するmRNAを同定し、特異的部位で切断します。次に、この切断が繰り返されることでmRNAが分解され、最終的にタンパク質の発現抑制が起こります。
miRNA解析
RNAポリメラーゼIIおよびIIIはともにmiRNAが含まれる遺伝子を転写して、長い一次転写産物(primiRNA)を生成し、これがRNase III型酵素のDroshaによってプロセシングされ70~90 bpのヘアピン構造(pre miRNA)が生じます。Pre miRNAヘアピンは細胞質に輸送され、RNase IIIタンパク質のDicerによって19~22ヌクレオチド長の短いmiRNA二本鎖までプロセシングされます。miRNA二本鎖はRNA誘導サイレンシング複合体(RISC)と呼ばれる 多重タンパク質ヌクレアーゼ複合体によって認識されますが、この時、二本鎖のうち一方の鎖であるガイド鎖がこのタンパク質複合体が相補的なmRNA転写産物を認識するように導きます。RISC-miRNA複合体は、しばしば不完全な配列相同性を示す領域で標的mRNAの3’UTRと相互作用し、いまだに十分に解明されていないメカニズムによりタンパク質合成を阻害します(図2)。

図2 miRNAの生合成と機能。RNAポリメラーゼIIおよびIIIによって生成したmicroRNA転写産物は、RNaseIII酵素のDrosha(核内)とDicer(細胞質内)によってプロセシングされて19~22ヌクレオチドのmiRNA2本鎖を生じる。この二本鎖のうちの一方の鎖はタンパク質発現を調節するRISC複合体に組み込まれる。
RNAi実験アプローチの選択
RNAi (RNA干渉)のプロセスはsiRNAまたはmiRNAによって緩和できます。これらはいずれもDicerと呼ばれる酵素によって細胞内でプロセシングされ、RISC(RNA誘導サイレンシング複合体)と呼ばれる複合体に組み込まれます。しがしながら、この2つにはわずかな違いがあります。
siRNAは、外来二本鎖RNAで、化学合成され、細胞に直接導入されるか、siRNAの前駆体である短いヘアピンRNA(shRNA)を発現するベクターを導入することによって細胞内で産生されます。一方、miRNAは一本鎖RNAで、より大きいRNA分子のイントロン内に存在する内在性ノンコーディングRNAです。しかしながら、shRNAから機能性siRNAへのプロセシングは、mRNAの安定性、翻訳、およびクロマチン構造を調節することによる遺伝子発現の細胞内調節の役割を持つ、ゲノムにコードされたmiRNAが自然にプロセシングされるのと同じ細胞内RNAi機構を介して行われます(Hutvagner and Zamore, 2002)。
siRNAとmiRNAの他の違いとしては、siRNAは一般的に動物におけるmRNAターゲットに完全かつ特異的に結合するのに対し、miRNA はそのペアリングが不完全であることから、多くの異なるmRNA配列の翻訳を阻害することが挙げられます。植物において、miRNAはより完全に相補的な配列を持つ傾向があり、単に翻訳を阻止するのとは対照的にmRNA開裂を誘導します。
siRNAとmiRNAはいずれもRNA誘導転写サイレンシング(RITS)と呼ばれるプロセスを介してエピジェネティクスに関与します。さらに、これらはいずれも遺伝子発現を制御する役割を担うため、治療用途の重要なターゲットです。
| siRNA | miRNA | |
| 発生 | 植物および下等動物で自然発生します。哺乳類で自然発生するかは解明されていません。 | 植物および動物で自然発生します。 |
| 構造 | 二本鎖 | 一本鎖 |
| 鎖長 | 21~22 nt | 19~25 nt |
| 標的miRNAに対する相補性 | 100%完全に一致するため、siRNAは特定の遺伝子をノックダウンし、マイナーなオフターゲットの例外が生じます。 | 正確ではないため、1つのmiRNAは最大数百のmRNAをターゲットとします。 |
| 生合成 | siRNAを発現する遺伝子と同じ遺伝子を調節します。 | miRNAを作製する目的の遺伝子によって発現されますが、miRNAを発現する遺伝子以外の遺伝子(mRNA)を調節します。 |
| 作用 | mRNAの切断 | mRNAの翻訳阻害 |
| 機能 | 抗体や細胞媒介性免疫を持たない植物および動物における遺伝子サイレンシングの監査役として機能 | 遺伝子(mRNA)の調節因子(阻害剤) |
| 用途 | siRNAは、ほぼ全ての分子生物学研究室で使用されている、遺伝子をノックダウンするための有用な実験室ツールです。いくつかのsiRNAが有望な治療薬として臨床試験中です。 | 薬剤ターゲットまたは薬剤自体としての治療用途が有望です。miRNAの発現レベルから、診断ツールおよびバイオマーカーツールとしての使用が有望視されています。 |
【無料ダウンロード】RNAiハンドブック
機能喪失研究のためのsiRNAやmiRNA用のツール、ワークフロー、アプリケーションがわかるハンドブックです。PDFファイルのダウンロードをご希望の方は、下記ボタンよりお申し込みください。
研究用にのみ使用できます。診断用には使用いただけません。